A estabilidade de proteínas é fundamental para o sucesso ou a falha no desenvolvimento de um produto biofarmacêutico. É um parâmetro importante para a produção, a fabricação, a formulação, o armazenamento a longo prazo, a entrega ao paciente e a eficácia. As proteínas altamente estáveis podem apresentar menos problemas durante o processo de fabricação, sua produção é mais econômica e têm mais chances de continuarem funcionais durante a formulação e o armazenamento sem agregação ou alteração química. Na abordagem “Qualidade por Design” (QbD) para o desenvolvimento biofarmacêutico, a caracterização de estabilidade faz parte da avaliação da “desenvolvibilidade” ou “farmacobilidade” de um possível fármaco candidato, assim como durante a fabricação e o desenvolvimento do processo. Os dados de estabilidade também são incorporados na caracterização da estrutura de ordem mais alta (HOS) e na “impressão digital” usada no suporte à fabricação, na biocomparabilidade e na biossimilaridade. A caracterização de HOS de proteína também é cada vez mais esperada em envios regulamentares de novos biofármacos e biossimilares.
Devido à natureza complexa de proteínas, as ferramentas biofísicas são importantes na caracterização completa de um produto biofarmacêutico. Há várias ferramentas biofísicas utilizadas para avaliar a estabilidade de proteínas, incluindo (mas não se limitando a) dicroísmo circular (CD), sistema de espalhamento de luz dinâmica e estática (DLS e SLS), cromatografia de exclusão por tamanho - espalhamento de luz de multiângulo (SEC-MALS), espectroscopia de infravermelho com transformada de Fourier (FTIR), ultrafiltração analítica (AUC), cromatografia de exclusão por tamanho (SEC), fluorescência de varredura diferencial (DSF), fluorescência intrínseca (IF) e calorimetria de varredura diferencial (DSC).
Embora todos esses ensaios biofísicos tenham um papel importante no desenvolvimento biofarmacêutico, a caracterização da estabilidade térmica pela DSC é fundamental. Em 2015, um artigo sobre técnicas biofísicas para a caracterização da estrutura de ordem mais alta de anticorpos monoclonais, Gokarn e outros afirmaram: “A DSC permanece como uma técnica incomparável para avaliar a estabilidade termodinâmica de proteínas em um determinado estado de tampão”[1].
O foco deste whitepaper é no uso da DSC para caracterizar a estabilidade térmica de produtos biofarmacêuticos de proteína (principalmente os anticorpos) e como uma ferramenta de caracterização de HOS para a comparabilidade dos produtos biofarmacêuticos (comparação de lote para lote, efeito de alterações no processo etc.) e para o desenvolvimento biossimilares.
A DSC é uma técnica de microcalorimetria usada para caracterizar a estabilidade térmica e conformacional de proteínas, ácidos nucleicos, lipídios e outros biopolímeros[2-7]. A DSC mede a capacidade térmica como uma função da temperatura. Os instrumentos da DSC utilizados para a caracterização de proteína descrita neste whitepaper são instrumentos de “compensação de energia”, com o biopolímero na solução, colocados em uma célula de amostra fixa e em uma célula de referência correspondente e preenchida com tampão. O sinal de capacidade de aquecimento (Cp) de uma célula de amostra é comparado com o sinal da célula de referência. À medida que a temperatura das células aumenta, a diferença de temperatura entre as células de referência e de amostra é medida continuamente e calibrada nas unidades de energia. A DSC é um ensaio da “degradação forçada”. Como a proteína está exposta à temperatura elevada, começa a se desdobrar e a Cp da proteína aumenta (Figura 1).
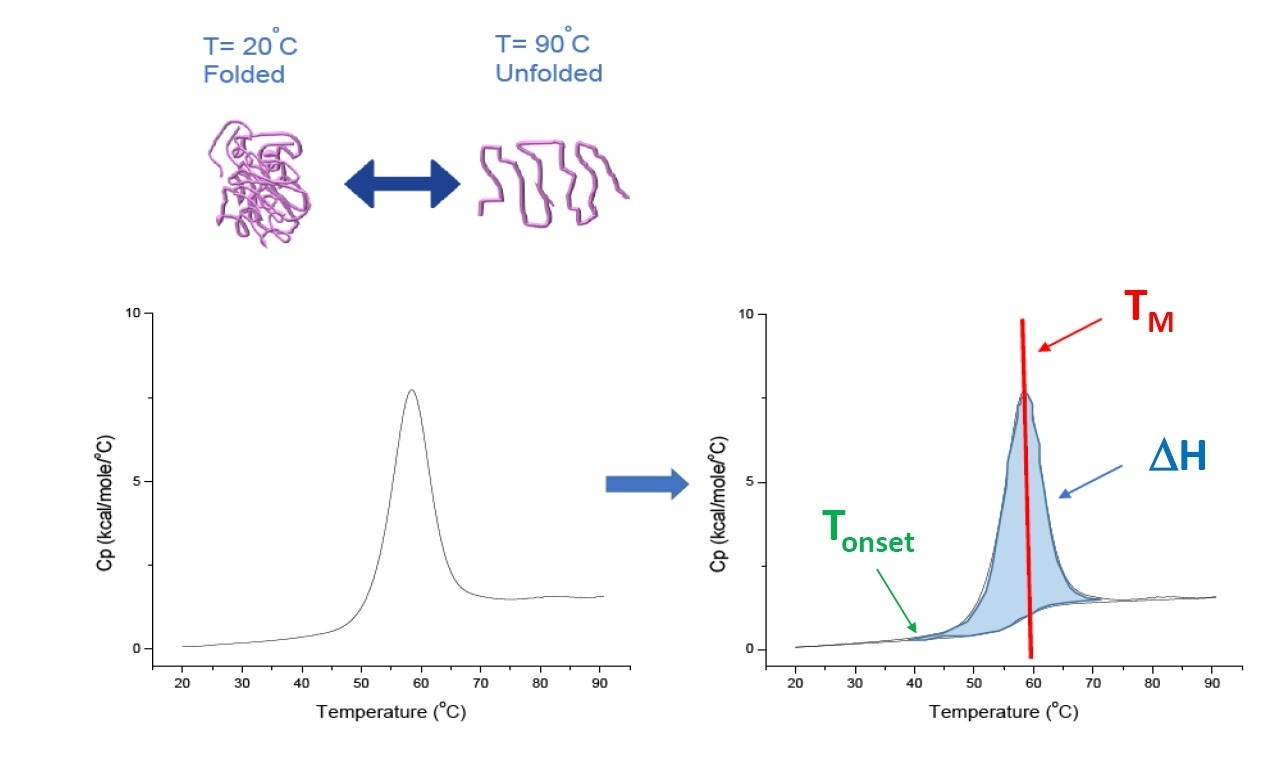
Figura 1: como funciona a DSC. A capacidade de aquecimento (Cp) muda conforme a desnaturação térmica de uma proteína. O experimento da DSC começa a uma temperatura em que a proteína é dobrada principalmente em sua conformação nativa. Com a temperatura elevada, em algum momento, a proteína vai começar a desdobrar/desnaturar (Tonset), e a Cp aumenta. Na temperatura em que 50% da proteína está na sua conformação nativa e 50% é desnaturada, a Cp atinge seu valor máximo - este é o ponto médio da transição térmica ou TM. Acima de TM, a proteína será desnaturada e, no fim do experimento da DSC, toda a proteína estará na sua conformação desdobrada. Parâmetros experimentais para a DSC incluem Tonset, TM e entalpia de desdobramento (ΔH)
A DSC mede diretamente a alteração da capacidade de aquecimento, sem precisar de fluorescência, rótulo ou sonda. Para uma proteína que desnatura de modo reversível, o ponto médio da transição térmica (TM), também chamado de temperatura de fusão ou desnaturação, é a temperatura na qual 50% da proteína está na sua conformação nativa (dobrada) e 50% está na sua conformação desnaturada. O TM é visto como o “pico” do termograma da DSC.
O TM é considerado como uma excelente indicação da estabilidade térmica. Quanto maior for o TM, maior será a estabilidade térmica da proteína. As proteínas de vários domínios (como anticorpos) geralmente têm mais de um pico em um termograma da DSC. Portanto, mais de um TM pode ser determinado em cada domínio (consulte a Figura 2).
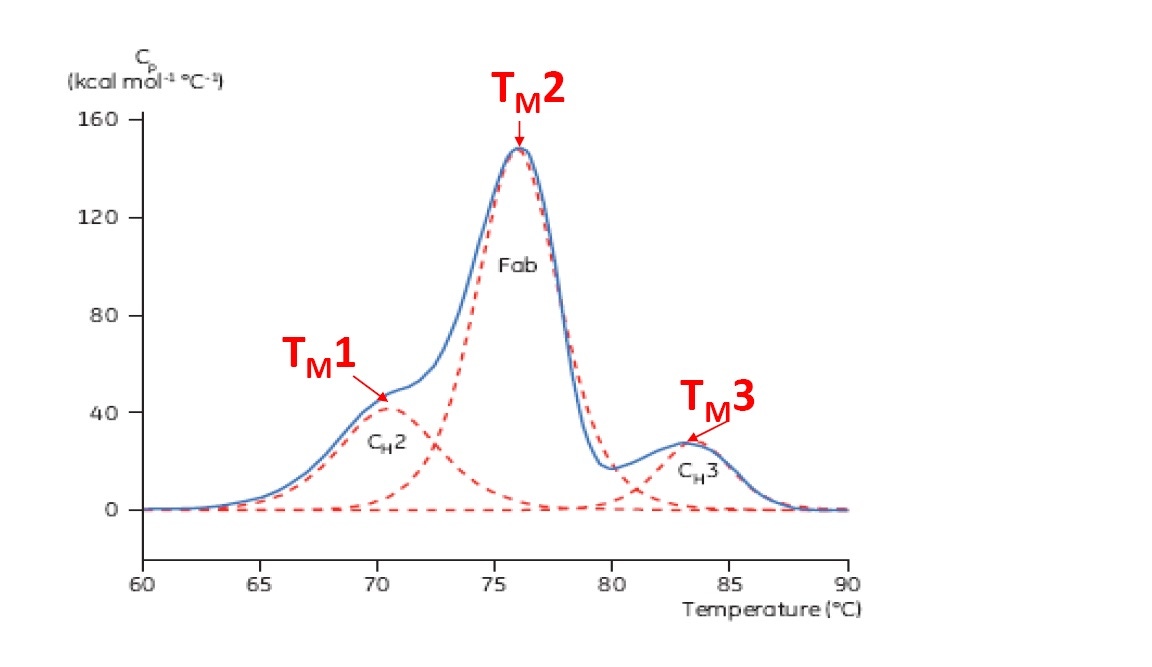
Figura 2: termograma representativo da DSC de um anticorpo monoclonal, com os domínios identificados CH2, Fab e CH3. As linhas vermelhas tracejadas são os picos de deconvolução da transição de cada domínio, com os três TMs indicados
A DSC oferece outros parâmetros úteis que podem ser usados para caracterizar e classificar a estabilidade de proteínas, incluindo a entalpia de desdobramento (ΔH), que é medida pela área abaixo da curva. O desdobramento de proteína é endotérmico, pois a entrada de energia é necessária para quebrar as ligas secundárias não covalentes que mantêm a proteína dobrada. A DSC também determina o Tonset (início do desdobramento), o ΔCp (alteração da capacidade de aquecimento do desdobramento) e o T1/2 (largura a 1/2 da altura do pico que indica a forma do termograma de desdobramento). A análise da DSC pode incluir a determinação de qualquer combinação desses parâmetros.
A desnaturação da maior parte das proteínas é irreversível. Elas tendem a agregar ou precipitar após a desnaturação térmica. O TM e outros parâmetros da análise da DSC de proteínas irreversivelmente desnaturadas não são os parâmetros termodinâmicos corretos. No entanto, a classificação do TM na DSC de uma desnaturação de proteína irreversível continua muito útil para a triagem de estabilidade de parâmetros qualitativos.
A Malvern Instruments oferece o sistema MicroCal VP-Capillary DSC[8,9] que é uma DSC automatizada projetada para a triagem do TM e a caracterização termodinâmica de proteínas e biopolímeros na solução.
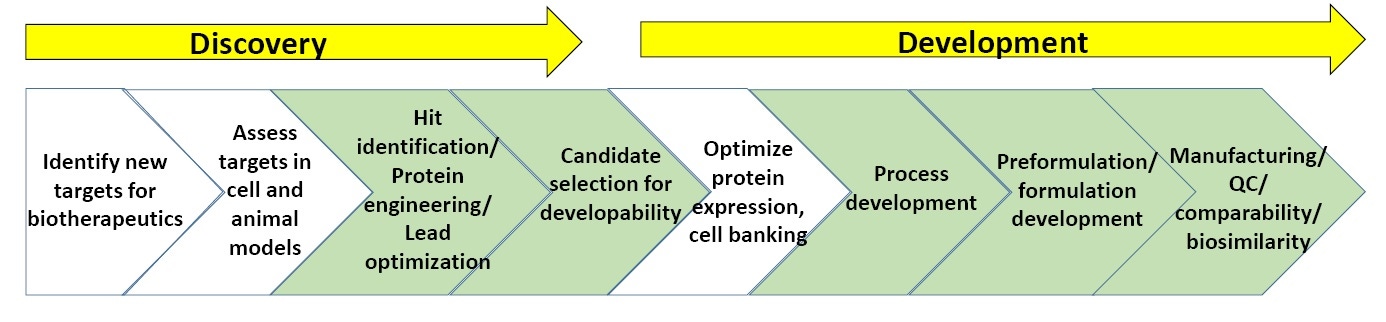
Figura 3: esquema geral de processos na descoberta e no desenvolvimento biofarmacêuticos
A Figura 3 mostra um esquema geral de processos na descoberta e no desenvolvimento biofarmacêuticos. As seções verdes são os pontos em que a caracterização biofísica, incluindo ensaios de estabilidade, é mais usada (no fim deste whitepaper, há uma lista de sugestões de leitura sobre a descoberta e o desenvolvimento biofarmacêuticos).
No desenvolvimento de um produto biofarmacêutico necessário, os cientistas inicialmente procuram biomoléculas que já demonstram elevada estabilidade durante a seleção de candidatos e podem precisar introduzir maior estabilidade por meio da engenharia de proteínas. Durante a purificação, a proteína é removida das condições em que é estável, dobrada corretamente e ativa. Por isso, é muito importante usar tampões, aditivos, purificação e condições de armazenamento que podem manter a estabilidade da proteína nesse processo.
Os fármacos de proteína subcutânea (SC) formulada devem ser estáveis e inalterados em concentrações de proteínas muito elevadas (às vezes, em torno de 100 mg/mL) no sistema de fechamento de recipiente (por exemplo, frasco ou seringa pré-preenchida) durante vários anos. Quando moléculas de proteínas são expostas a excesso de calor, produtos químicos, alterações no pH, pressão, mistura e alta concentração, durante a produção e a formulação biofarmacêuticas, a conformação da proteína pode favorecer a proteína desnaturada (desdobrada). As proteínas na solução também são suscetíveis a modificações, como desamidação e oxidação que também levarão a proteínas desnaturadas e inativas. No caso de um produto biofarmacêutico de proteína, a desnaturação e outras modificações podem resultar na formação de agregações que têm eficácia reduzida ou são ineficazes como fármaco. A agregação de proteína pode resultar em uma resposta imunogênica possivelmente fatal em um paciente. O uso de uma proteína estável como base de um produto biofarmacêutico resulta em uma produção mais econômica e um fármaco eficiente e seguro.
A sequência de aminoácidos ou a estrutura primária (1°) de uma proteína é o componente mais básico da cadeia polipeptídica e da estrutura de proteínas. Além disso, é importante entender e caracterizar a estrutura tridimensional da proteína também denominada como estrutura de ordem mais alta (HOS). Há três níveis de HOS de proteína: o secundário (2°) refere-se aos padrões de desdobramento local da estrutura primária de uma proteína, incluindo α-helix, β-shee, curvas e bobinas aleatórias; o terciário (3°) é a estrutura 3D final de uma proteína decorrente de uma matriz de elementos estruturais secundários; o quaternário (4°) descreve as estruturas que envolvem a interação de duas ou mais cadeias polipeptídicas idênticas ou diferentes.
A DSC é uma medida da estabilidade conformacional e das alterações nas estruturas terciária e quaternária que ocorrem quando uma proteína é desnaturada termicamente, fornecendo uma indicação de como os fatores intrínsecos e extrínsecos afetam a estabilidade de uma proteína. A DSC é considerada como o melhor e o mais quantitativo ensaio para a estabilidade térmica utilizada na caracterização de proteínas biofarmacêuticas, como um indicador de estabilidade em longo prazo[1,10-14]. O TM da DSC é um parâmetro usado frequentemente para classificar a estabilidade na seleção de candidatos (desenvolvibilidade), triagem de formulação e desenvolvimento do processo, com proteínas mais estáveis que têm um TM mais alto. Entalpia (ΔH), Tonset, T1/2 e ΔCp da DSC também são usados na estabilidade de classificação, na validação de dados da DSC, na análise quantitativa do desdobramento de proteínas e em uma “impressão digital”[10-14] de estrutura de ordem mais alta.
A análise da DSC de uma proteína nas condições de solução definidas é quantitativa e reproduzível caso a proteína seja a mesma ou muito semelhante (Figura 4). Ou seja, os termogramas da DSC terão um padrão reproduzível e os parâmetros (incluindo TM, ΔH e Tonset) estarão em uma faixa[12-14] aceitável. Caso os termogramas comparados sejam diferentes e os parâmetros de ajuste da DSC sejam alterados, isso sugere que houve um evento, como desdobramento, degradação, agregação, alteração em solvente, modificação pós-traducional de proteína ou outra alteração de estrutura de ordem mais alta, o que afeta a estabilidade conformacional.
Os dados reproduzíveis e quantitativos tornam a DSC um ensaio importante na comparabilidade e na análise de HOS para a avaliação de produtos durante a fabricação (incluindo comparabilidade de lote para lote e de local para local), a comparação de variantes de proteína e de produtos modificados (como mudanças estruturais devido à glicosilação e a oxidação) e a biossimilaridade. Os dados da DSC também são usados em documentos regulamentares de suporte como uma caracterização de HOS para submissões de novos fármacos e biossimilares. Em uma pesquisa de cientistas no setor biofarmacêutico, a DSC foi classificada como uma ferramenta biofísica de HOS “muito útil” a “extremamente útil” para a seleção de candidatos, o desenvolvimento de formulação, a caracterização de produtos, a comparabilidade, o desenvolvimento de processos e a biossimilaridade[15].
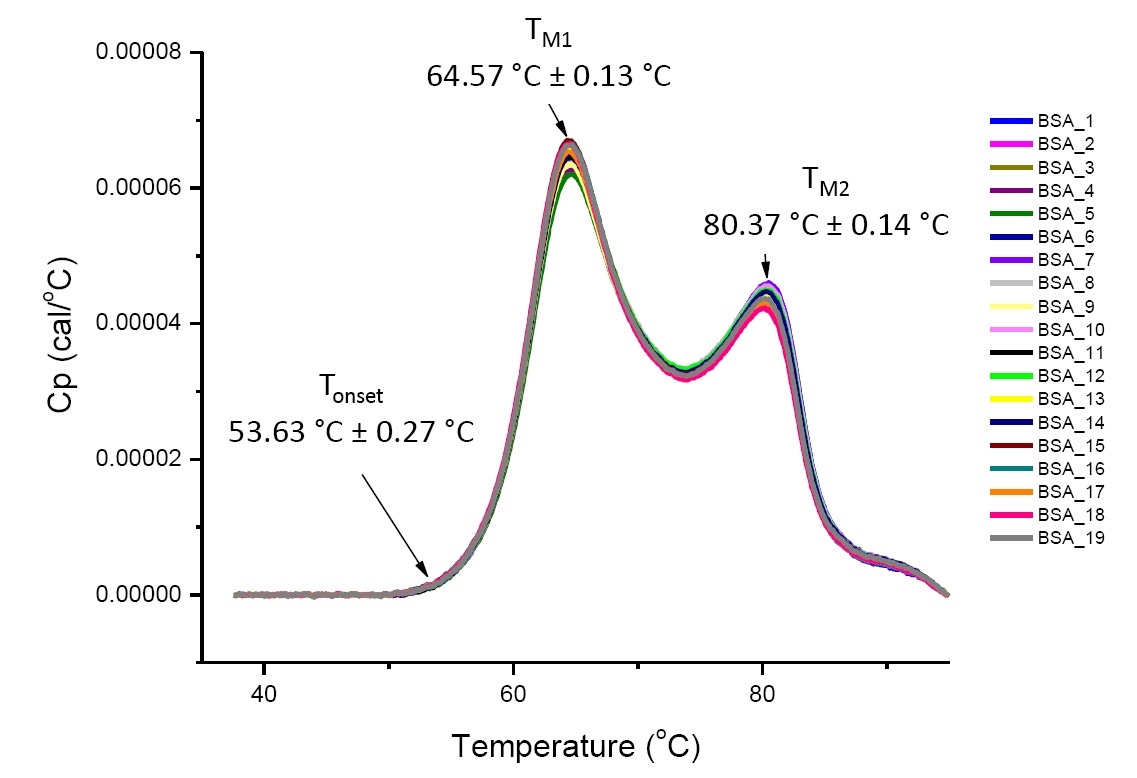
Figura 4: dezenove termogramas da DSC de albumina sérica bovina (Sigma A1933, purificada cromatograficamente) em PBS. Dados da DSC mostrados após a normalização da velocidade de varredura, subtração de tampão-tampão e subtração da linha de base de integração. O valor médio e os desvios padrão de Tonset, TM1 e TM2 são mostrados
Para proteínas de vários domínios, como anticorpos, os termogramas da DSC têm mais de uma transição de desdobramento (conforme a Figura 2). A DSC é capaz de caracterizar e quantificar os diferentes domínios e determinar os TMs individuais para cada transição. Os valores de TM são representados pelos picos do termograma e podem ser determinados a partir dos dados da DSC, sem a necessidade de análises de dados complexos. Os ensaios biofísicos alternativos que podem o determinar o TM (como CD, IF e DSF) só podem detectar o primeiro TM (que ocorre na temperatura mais baixa) ou o TM “mais dominante” para proteínas de vários domínios. A extração de mais de um TM de dados espectroscópicos e fluorescentes requer o ajuste de dados complexos e talvez eles não sejam reproduzíveis.
A DSC não exige mais amostras de proteínas por varredura e pode ter menor rendimento em relação a outros ensaios de triagem do TM. Caso a amostra seja limitada, uma solução recomendável seria efetuar a classificação do TM inicial com DSF ou IF e, depois, escolher diversas amostras para validar o TM com a DSC). É importante para validar os resultados do TM com a DSC e não depender só da fluorescência ou da espectroscopia para o cálculo do TM para avaliar estabilidade. É comum em ensaios de fluorescência para ver os artefatos que interferem com a saída, e os resultados do TM podem aparecer para passar para um valor maior (ou menor) devido a esses artefatos. Algumas proteínas e condições de tampão não são compatíveis com a fluorescência, que pode complicar o cálculo das diferenças do TM. Por fim, a fluorescência e a espectroscopia não podem determinar a entalpia calorimétrica e outros parâmetros termodinâmicos, enquanto a DSC é capaz de fornecer todas essas informações como um “conjunto de dados” de estabilidade térmica.
A DSC é considerada pelo setor biofarmacêutico como o ensaio de estabilidade térmica “padrão de ouro” porque a técnica:
Mede as alterações de calor associadas ao desdobramento de proteínas
É uma medida direta do desdobramento de proteínas, por isso, não é necessário rótulo ou identificador. Isso significa que não há artefatos de detecção possíveis que são normalmente encontrados em ensaios fluorescentes ou espectroscópicos
Mede proteínas nativas em solução e pode ser usada com praticamente todos os tampões e aditivos que estão em uso comum na purificação biofarmacêutica e no desenvolvimento de formulação. Alguns desses tampões e aditivos não são compatíveis com fluorescência ou espectroscopia
É fácil de usar na configuração experimental
Tem controle de temperatura de alta precisão, com uma faixa de temperatura de funcionamento de até 130°C, por isso, pode detectar a maioria das transições de TM alto. Outros ensaios de triagem de TM podem aquecer amostras em até 100°C (ou mais baixa)
É um ensaio de “degradação forçada”, portanto, não requer o armazenamento da proteína no tampão antes da análise. SEC-HPLC e DLS geralmente exigem amostras de incubação no tampão a uma temperatura elevada para detectar alterações na conformação
Tem saída de dados simples e o software de análise de dados integrados
Pode ser usada para resolver transições de desdobramento separado e caracterizar proteínas com vários domínios e complexos de proteínas, além de proteínas de domínio único simples
Tem informações importantes, como dados termodinâmicos, estabilidade conformacional e determinação de TM
Pode ser usada como principal ensaio para a caracterização da estabilidade térmica bioterapêutica e com outras ferramentas de triagem biofísica que são ortogonais ou complementares e/ou para validar outros dados
Está disponível com a automação de alto rendimento (sistema MicroCal VP-Capillary DSC) para triagem rápida da estabilidade térmica
Comparabilidade biofarmacêutica
Anticorpos monoclonais e outros biofármacos são proteínas complexas representadas por células de mamíferos ou bacterianas. Cada proteína é exclusiva e requer uma extensa pesquisa, caracterização e otimização para transformá-la em um fármaco. Como parte da descoberta e do desenvolvimento inicial, são definidas as propriedades desejadas do fármaco de proteína, que incluem pureza, potência e dosagem. Os ensaios bioquímicos, biofísicos e biológicos são desenvolvidos para estudar essas características e são otimizados durante o desenvolvimento do produto.
A caracterização de proteína detalhada e o HOS (incluindo estabilidade) são feitos por cada processo para definir os principais atributos de qualidade da proteína e os parâmetros de processos fundamentais, que são necessários para controlar e validar os processos em todo o ciclo de vida do produto. Na abordagem “Qualidade por Design” (QbD) para o desenvolvimento biofarmacêutico, os principais atributos de qualidade e a caracterização de estabilidade fazem parte da avaliação do fármaco durante o desenvolvimento do processo e o suporte[16,17] à fabricação.
Durante o processamento de um fármaco de proteína, a proteína está exposta a condições diferentes, incluindo:
Soluções diferentes, como tampão, pH e sais variados
Aditivos de formulação (excipientes)
Diversas concentrações de proteínas
Ciclos repetidos de congelamento/descongelamento, aumento das pressões e mistura/agitação
Diferentes superfícies de material com as quais a proteína tem contato (bombas, membranas de ultrafiltração/diafiltração, mídia de cromatografia etc.)
Exposição a oxidantes, proteases, mídia de crescimento de célula, variantes de produtos etc.
Qualquer uma dessas condições pode interromper as intensidades e as interações que mantêm uma proteína em sua conformação dobrada nativa, o que resulta em uma proteína desnaturada ou inativa. Pode ocorrer a desnaturação química em virtude de oxidação, desamidação, alterações na glicosilação ou outras modificações pós-traducionais.
As proteínas desnaturadas frequentemente formam agregados, que é um ponto importante para o desenvolvimento biofarmacêutico. Os agregados são estados associados do monômero de proteína, e a formação de agregados pode ser reversível ou irreversível, com tamanhos que variam de um dímero de proteína a partículas grandes que são visíveis a olho nu. No mínimo, a agregação de proteína reduz a eficácia e a potência dos fármacos e aumenta o custo da produção. A maior preocupação com agregados de proteína e partículas é a sua capacidade de originar respostas imunogênicas, o que pode resultar na morte do paciente em casos extremos. Por causa desse problema, a agregação de proteína recebeu uma análise minuciosa no setor biofarmacêutico. Isso inclui detecção de partícula/agregação, quantificação e caracterização durante a produção e a formulação, além de uma busca de soluções para reduzir ou eliminar a agregação de proteína em produtos biofarmacêuticos.
É importante para demonstrar que o fármaco de proteína fabricada é semelhante em estrutura, estabilidade, distribuição de tamanho e ensaios bioquímicos e funcionais para:
A mesma proteína fabricada em lotes anteriores e o lote de proteína de referência
A mesma proteína fabricada em diferentes locais
A mesma proteína fabricada com um processo modificado de upstream ou downstream
Intensificação de upstream ou processo de downstream
A mesma proteína em uma formulação diferente
Ensaios bioquímicos, biofísicos, de HSO e funcionais para comparabilidade (ou biocomparabilidade) demonstram que o produto de proteína fabricada é “muito semelhante” nos principais atributos de qualidade, quando comparado com uma proteína de referência. É essencial que o HOS dos fármacos candidatos de proteína seja analisado com cuidado em cada estágio de desenvolvimento.
As alterações no processo de fabricação de um produto biofarmacêutico podem ocorrer durante o desenvolvimento do processo e após a aprovação do fármaco. Estes são os motivos das alterações:
Melhoria do processo de fabricação (por exemplo, rendimento do produto, custo reduzido)
Aumento na escala
Mudança do local de fabricação
Alterações regulamentares e de conformidade
Melhoria na estabilidade do produto
Quando mudanças significativas são feitas no processo de fabricação, um exercício de comparabilidade para avaliar o impacto das alterações nos principais atributos de qualidade, a segurança e a eficácia do produto biofarmacêutico devem ser realizadas. A demonstração da comparabilidade não significa necessariamente que os principais atributos de qualidade do produto antes e após a alteração sejam idênticos, mas que sejam muito semelhantes. As alterações de fabricação não devem ter impacto negativo na qualidade do produto.
A comparabilidade é a principal preocupação para a terapêutica proteica e é abordada por várias agências [18,19,20] reguladoras internacionais. Nenhum método analítico pode ser usado para avaliação de comparabilidade de fármacos de proteína. Os estudos de HSO e comparabilidade são (mas não se limitam a): DSC, DLS, fluorescência, CD, AUC, SEC, espectroscopia Raman, Análise de Rastreamento de Nanopartículas (NanoSight da Malvern Instruments), medição de massa ressonante (Archimedes da Malvern Instruments) e microscopia. A bioatividade e eficácia de proteína são também controladas com bioensaios, calorimetria de titulação isotérmica (ITC) e ressonância plasmônica superficial (SPR).
Os dados fornecidos por cada método biofísico são específicos e, quando avaliados em conjunto, viabilizam informações estruturais sobre a caracterização do HOS e a comparabilidade. O “mapa” do HOS ajuda a garantir a consistência na estrutura de proteínas em relação à intensificação durante o ciclo de desenvolvimento do processo, a comparabilidade de lote para lote, quando os processos são transferidos para novos locais de fabricação e quando a produção é modificada. As informações da caracterização do HOS também são essenciais para avaliar os biossimilares.
É importante observar que os testes e a instauração da comparabilidade durante um processo de fabricação (ou alteração) não são os mesmos que os testes executados durante o desenvolvimento de um fármaco biossimilar - o desenvolvimento de biossimilares é um processo muito mais complexo. Como um biossimilar geralmente não é produzido pela mesma empresa que desenvolveu o inovador (também denominado produto parental ou de referência), o desenvolvimento de biossimilares precisa de “engenharia reversa” para estabelecer os novos processos de upstream e downstream. O fabricante do biossimilar deve combinar a qualidade do produto (biossimilaridade) na faixa estreita do produto comercial do inovador, o que exige mais evidência biofísica e bioquímica para demonstrar biossimilaridade do que é necessário para mostrar a comparabilidade durante a fabricação.
Como a DSC fornece informações sobre a estabilidade térmica de uma proteína em diferentes condições de solvente e os resultados da DSC são reproduzíveis (Figura 4), a DSC é incluída com frequência nos estudos de comparabilidade para mostrar que o processo não resulta em uma alteração na estabilidade do produto, e que os produtos fabricados a partir de diferentes lotes e/ou locais de fabricação são muito parecidos.
Jiang e Nahri[21] analisaram diversas técnicas biofísicas de comparabilidade durante a fabricação e o desenvolvimento do processo. Mencionaram o uso da DSC como uma das ferramentas de caracterização biofísica utilizadas na comparabilidade de um fármaco de anticorpo monoclonal representado por duas linhas celulares diferentes (linhas celulares 1 e 2), que também apresentaram uma alteração no processo de produção (processos 2pA e 2pB). Os três produtos de proteína resultantes foram avaliados por FTIR (para determinar a estrutura secundária), CD de UV próximo e fluorescência intrínseca (para comparar a estrutura terciária), fluorescência e ligação de ANS (para comparar a hidrofobicidade de superfície), DLS (para comparar as propriedades hidrodinâmicas e a distribuição do tamanho das proteínas) e DSC (para comparar a estabilidade térmica e a solubilidade).
Os resultados deste conjunto de ensaios biofísicos mostraram que as estruturas secundária e terciária das três amostras foram semelhantes. Os resultados da DSC indicaram que a estabilidade térmica da proteína na linha celular 2pB aumentou de forma significativa em relação à amostra 2pA e à amostra da linha celular 1. As amostras 2pA e da linha celular 1 também pareceram mais heterogêneas, conforme a DSC. Normalmente, as transições 2 a 3 são vistas para os anticorpos (veja a Figura 2 para obter um exemplo de um termograma “típico” da DSC de anticorpo). A presença de um número mais alto das transições térmicas sobrepostas, de acordo com a análise da DSC dessas amostras, indicou a heterogeneidade da amostra. Os resultados da DSC mostraram que a alteração do processo melhorou a homogeneidade e a estabilidade do anticorpo monoclonal de interesse. Com base nos dados de comparabilidade dos ensaios biofísico, bioquímico e funcional, abordados no artigo, a linha celular 2 e o processo 2pB foram considerados para produzir a proteína[21] mais estável.
Um segundo exemplo de Jiang e Nahri[21] utilizou a DSC para verificar a comparabilidade de um produto de proteína produzido em diversos locais de fabricação. A Proteína Y era composta de duas cadeias polipeptídicas monoméricas, ligadas por dissulfeto através da região Fc da molécula. Durante o curso de seu desenvolvimento, a Proteína Y foi fabricada em diferentes locais. Para verificar se a proteína presente nesses diferentes lotes era semelhante na conformação nativa, nas estruturas secundária e terciária, na estabilidade térmica e na distribuição de tamanho, quatro diferentes amostras da Proteína Y (que representam três amostras fabricadas em diferentes locais) e a proteína de referência padrão foram analisadas por FTIR, CD de UV distante, CD de UV próximo, fluorescência, DSC e AUC.
As varreduras de DSC do padrão de referência e 3 amostras de locais de fabricação diferentes mostraram duas transições térmicas para cada amostra, e todos os quatro perfis da DSC foram idênticos na variabilidade experimental. Isso mostrou que não havia diferença na estabilidade térmica das amostras de proteína e que todas foram dobradas na conformação nativa. Os resultados combinados de caracterização biofísica, incluindo os produzidos pela DSC, demonstraram que as quatro amostras da Proteína Y eram semelhantes, tinham as estruturas secundárias e terciárias apropriadas e eram homogêneas[21].
Se os termogramas da DSC não fossem idênticos, haveria uma alteração na modificação pós-traducional ou uma desnaturação química na proteína, relacionada ao processo ou à formulação. Arthur e outros[22] demonstraram a sensibilidade da DSC na detecção das alterações do HOS na estabilidade decorrente da oxidação, uma passagem de degradação química comum. Se um produto biofarmacêutico for submetido à oxidação durante a purificação ou o armazenamento na formulação final, isso poderá afetar negativamente a eficácia e aumentará a probabilidade da formação de agregação de proteína. Neste estudo, os produtos de proteína das três classes estruturais foram avaliados em vários níveis de oxidação. Cada proteína mostrou um decréscimo linear no TM como uma função da oxidação de metionina; os autores também observaram diferenças na taxa de alteração do TM, além de diferenças na estabilidade de TM de domínio, nas classes[22] estruturais. Em contrapartida, o CD de UV próximo e a espectroscopia de fluorescência eram muito menos sensíveis a alterações conformacionais induzidas por oxidação. A DSC é um método de caracterização estrutural mais adequado para monitorar a oxidação de proteína, em relação a esses métodos[22] espectroscópicos. Para uma proteína no estudo (IgG2B), as alterações no TM foram detectadas pela DSC antes de uma perda na potência relativa, o que demonstra que a DSC é o principal indicador de redução da ligação ao antígeno. As alterações detectáveis na metionina oxidada pela espectrometria de massa (MS) ocorreram nos níveis de oxidação abaixo dos níveis com um impacto funcional ou conformacional detectável. Usando os deslocamentos do TM na DSC com a MS e os métodos de potência, a relação entre uma modificação estrutural primária, as alterações no HOS e na estabilidade conformacional e o impacto funcional pode ser caracterizada de forma confiável.
Morar-Mitrica e outros[12] descrevem vários estudos de caso que incorporam a DSC no HOS e estudos de comparabilidade de produtos biofarmacêuticos, incluindo a caracterização da oxidação de anticorpo monoclonal por deslocamentos de TM observáveis (semelhantes aos descritos em [22]), perfis de termograma da DSC reproduzível e o uso de TM, T1/2, Tonset e ∆H para estudos de comparabilidade de produtos biofarmacêuticos, além da caracterização da DSC estendida de anticorpos monoclonais glicosilados com diferentes níveis de glicosilação (uma modificação pós-traducional comum).
Shahrokl e outros[23] mostram como a DSC é útil em estudos de comparabilidade como parte da aplicação de licença biológica e mencionam a facilidade de uso da DSC e a simplicidade da saída de dados. Os autores descrevem como a DSC foi usada na comparabilidade de uma glicoproteína 51 kDa antes e depois de uma alteração no seu processo de fabricação. A alteração do processo foi implementada para aumentar o rendimento do produto e remover materiais de origem animal no meio da cultura celular. Usando a DSC, os autores avaliaram três lotes da glicoproteína que foram produzidos com cada processo de fabricação, observaram que os termogramas da DSC foram sobrepostos e mostraram uma única transição a 60,7°C +/- 0,1°C. Os resultados da DSC mostraram que a alteração de fabricação não afetou a estabilidade conformacional da glicoproteína[23].
Lubiniecki e outros[24] avaliaram o impacto das alterações de fabricação na função e estrutura de anticorpos durante o curso de desenvolvimento do produto, ao realizar três estudos de comparabilidade para dois candidatos diferentes de anticorpo monoclonal IgG1 (anticorpo A e anticorpo B). Dois dos estudos de comparabilidade incorporaram a DSC como uma das ferramentas analíticas bioquímicas e biofísicas. Um dos estudos verificou a intensificação do processo e a transferência para o local de fabricação, junto com a alteração de uma forma de dosagem liofilizada para líquida. Os termogramas da DSC para as formulações liofilizadas e líquidas do anticorpo A e anticorpo B mostraram uma excelente correlação[24]. A DSC, usada em conjunto com MS, CD, SEC e outros ensaios, mostrou que a alteração de fabricação não resultou na estrutura ou função dos anticorpos.
O terceiro estudo de comparabilidade avaliou a formulação líquida do anticorpo nos frascos[24] ou nas seringas pré-preenchidas. Os resultados de DSC, CD, SEC e outros ensaios mostraram uma estrutura molecular, uma atividade biológica e perfis de degradação semelhantes. Uma exceção notável foi um pequeno aumento (mas significativo) nos níveis de partículas subvisíveis em seringas[24] pré-preenchidas.
Um produto biossimilar, também chamado de “follow-on biologic” (biológico de prosseguimento) ou “subsequent entry biologic” (biológico de entrada subsequente), é um biofarmacêutico que foi aprovado por uma agência reguladora com base na sua alta similaridade em um produto biológico aprovado anteriormente, conhecido como produto de referência (também denominado produto parental ou inovador). Os biossimilares estão disponíveis globalmente há vários anos[25-29]. No momento em que este whitepaper foi escrito (outubro de 2016), o FDA dos Estados Unidos aprovou quatro biossimilares, e o EMEA aprovou 19 biossimilares.
Observe que o desenvolvimento de um biossimilar não é igual ao desenvolvimento de um fármaco genérico de pequenas moléculas. Os fármacos de pequenas moléculas são produtos químicos, e a síntese desses fármacos é um processo controlado. Como os produtos biofarmacêuticos normalmente são proteínas, existe um certo grau de variação de proteína, até mesmo entre diferentes lotes do mesmo produto, devido à variabilidade inerente do sistema de expressão biológica e ao processo de fabricação. Essas variações podem incluir diferenças na modificação pós-traducional e no HOS. Por causa dos altos pesos moleculares das proteínas, das estruturas complexas e das variações naturais, os biossimilares não são tão simples de produzir quanto os genéricos. Um biossimilar não é uma cópia exata do produto de referência. Por isso, é caracterizado como “semelhante” ou “muito semelhante” ao produto de referência.
O desenvolvimento de um biossimilar envolve comparações físico-químicas, analíticas e funcionais da proteína de referência e do biossimilar. Estes ensaios são complementados por dados comparativos clínicos e não clínicos para estabelecer eficiência e segurança equivalentes. Antes da aprovação, deve ser verificado se o biossimilar não tem diferenças clinicamente significativas em termos de segurança e eficácia do produto de referência. Apenas pequenas diferenças em componentes clinicamente inativos são permitidas em produtos biossimilares. O biossimilar deve trabalhar exatamente da mesma forma que o produto de referência para as mesmas indicações aprovadas.
A agências reguladoras avaliam os biossimilares com base no nível de semelhança com seus produtos de referência. Devido à complexidade dos produtos biofarmacêuticos, não é provável que dois fabricantes diferentes produzam dois produtos idênticos, mesmo se forem usados sistemas de expressão de host, processos e tecnologias equivalentes. Portanto, os produtores de biossimilares dependem de análises e estudos de comparabilidade do HOS, conforme especificado anteriormente[30,31]. A necessidade de mais informações analíticas e a busca de cronogramas reduzidos no desenvolvimento de biossimilares exigem a demonstração da comparabilidade com a molécula de referência em cada estágio, principalmente durante a fabricação.
Os biossimilares devem ter “engenharia reversa” para combinar o produto de referência da melhor maneira possível. A caracterização analítica de um biossimilar inclui uma avaliação na estrutura de ordem mais alta e principal (secundária, terciária e quaternária), atividade biológica, análise do produto e impurezas do processo. A DSC é usada como um ensaio biofísico do HOS para mostrar que um biossimilar tem uma “impressão digital” do perfil da DSC altamente semelhante, TM e Tonset similares, além de outros parâmetros termodinâmicos, em relação ao produto de referência.
Três dos quatro biossimilares aprovados pelo FDA dos Estados Unidos (desde outubro de 2016) incluíram a DSC como um dos ensaios do HOS no arquivamento do FDA:
Erelzi (da Sandoz, Inc.) é um biossimilar para Enbrel (etanercept)[32] da Amgen
Inflectra (da Celltrion), conhecido como Remsima em outros mercados, é um biossimilar para Remicade (infliximab) [33,34] da Janssen
Amjevita (da Amgen) é um biossimilar para Humira (adalimumab)[35,36] da Abbvie
Sinha-Datta e outros[37] demonstraram o uso da DSC (MicroCal VP-Capillary DSC) e da ressonância plasmônica superficial (SPR) como ferramentas de análise para comparar dois anticorpos monoclonais terapêuticos (mAb1-i e mAb2-i) com seus biossimilares (mAb1-B e mAb2-B1, B2, B3), com base na estabilidade térmica, cinética e afinidade. Eles observaram que os biossimilares eram altamente semelhantes às suas amostras de referência, em relação à biofunção dos resultados da SPR. A DSC foi usada como outra confirmação da biossimilaridade entre os produtos parentais e biossimilares. A análise da DSC mostrou uma excelente similaridade estrutural para os anticorpos parentais e biossimilares, com o principal TM a 84,1°C (para mAb1) e 72,8°C (para mAb2).
As informações apresentadas neste whitepaper demonstra a importância e o valor da incorporação da DSC como uma estabilidade biofísica e um ensaio do HOS para a caracterização da biossimilaridade e comparabilidade biofarmacêutica. Ao usar os resultados da DSC com outros ensaios biofísicos e bioquímicos, as empresas biofarmacêuticas poderão tomar decisões adequadas sobre a estabilidade de proteínas e a comparabilidade durante a produção, além de garantir que cada lote de proteína seja muito semelhante ao lote de referência e que qualquer alteração no processo ou na fabricação não comprometa a estabilidade conformacional de proteínas. A DSC também está incluída como um ensaio do HOS no desenvolvimento biossimilar para ajudar a demonstrar que o biossimilar é “muito semelhante” ao inovador.
Analytical Techniques for Biopharmaceutical Development, R. Rodriguez-Diaz, T. Wehr, S. Tuck (eds.), Taylor & Francis, New York, NY, EUA (2005).
Biophysical Characterization of Proteins in Developing Biopharmaceuticals, D.J. Houde, S.A. Berkowitz (eds.), Elsevier, Amsterdã, Holanda (2015).
Biophysical Methods for Biotherapeutics: Discovery and Development Applications, T.K. Das (ed.) John Wiley & Sons, Hoboken, NJ, EUA (2014).
Biophysics for Therapeutic Protein Development, L.O. Nahri (ed.), Springer, New York, NY, EUA (2013).
State-of-the-Art and Emerging Technologies for Therapeutic Monoclonal Antibody Characterization Volume 1. Monoclonal Antibody Therapeutics: Structure, Function, and Regulatory Space, J.E. Schiel, D.D. Davis, O.V. Borisov (eds.), ACS Symposium Series Vol 1176 (2014). doi: 10.1021/bk-2014-1176.
State-of-the-Art and Emerging Technologies for Therapeutic Monoclonal Antibody Characterization Volume 2. Biopharmaceutical Characterization: The NISTmAb Case Study, J.E. Schiel, D.D. Davis, O.V. Borisov (eds.), ACS Symposium Series Vol 1201 (2015). doi: 10.1021/bk-2015-1201.
State-of-the-Art and Emerging Technologies for Therapeutic Monoclonal Antibody Characterization Volume 3. Defining the Next Generation of Analytical and Biophysical Techniques, J.E. Schiel, D.D. Davis, O.V. Borisov (eds.), ACS Symposium Series Vol 1202 (2015) doi: 10.1021/bk-2015-1202.
1. Gokarn, Y., Agarwal, S., Arthur, K. e outros, Biophysical Techniques for Characterizing the Higher Order Structure and Interactions of Monoclonal Antibodies, in: State-of-the-Art and Emerging Technologies for Therapeutic Monoclonal Antibody Characterization Volume 2. Biopharmaceutical Characterization: The NISTmAb Case Study. J.E. Schiel, D.D. Davis, O.V. Borisov (eds.), ACS Symposium Series Vol 1201, American Chemical Society, Washington DC, EUA, páginas 285-327 (2015).
2. Cooper, A., Nutley, M. A. e Wadood, A., Differential Scanning Calorimetry, in: Protein-Ligand Interactions: Hydrodynamics and Calorimetry, A Practical Approach, S.E. Harding, B.Z. Choudry (eds). Oxford University Press, Oxford, Reino Unido, p. 287-318 (2001).
3. Whitepaper da Malvern Instruments “Differential Scanning Calorimetry (DSC) Theory and Practice” http://www.malvern.com/en/support/resourcecenter/Whitepapers/WP140701-dsc-theory-and-practice.aspx.
4. Bruylants, G., Wouters, J. e Michaux, C., Current Med. Chem. 12, 2011-2020 (2005) doi: 10.2174/0929867054546564.
5. Jelesarov, I. e Bosshard. H.R., J. Mol. Recognit. 12, 3-18 (1999) doi: 10.1002/ (SICI)1099 1352(199901/02)12:1<3:AID-JMR441>3.0.CO; 2-6.
6. Choi, M.H. e Prenner, E.J., J. Pharm. Bioallied Sci. 3, 39-59 (2011) doi: 10.4103/0975-7406.76463.
7. Johnson, C.M., Arch. Biochem. Biophys. 531, 100-109 (2013) doi: 10.1016/j.abb.2012.09.008.
8. Plotnikov, V., Rochalski, A., Brandts, M., Brandts, J.F., Williston, S., Frasca, V. e Lin, L.N., Assay Drug Devel. Technol. 1, 83-90 (2004) doi:10.1089/154065802761001338.
9. www.malvern.com.br
10. Demarest, S.J. e Frasca. V., Differential Scanning Calorimetry in the Biopharmaceutical Sciences, in: Biophysical Characterization of Proteins in Developing Biopharmaceuticals, D.J. Houde, S.A. Berkowitz (eds.), Elsevier, Amsterdã, Holanda, p. 287-306 (2015).
11. Remmele, R.L., Microcalorimetric Approaches to Biopharmaceutical Development, in: Analytical Techniques for Biopharmaceutical Development, R. Rodriguez-Diaz, T. Wehr, S. Tuck (eds.), Taylor & Francis, Nova York, NY, EUA, p. 327-381 (2005).
12. Morar-Mitrica, S., Nesta, D. e Crofts, G., BioPharm Asia 2, 46-55 (2013).
13. Kirkitadze, M., Hu, J., Tang, M. e Carpick, B., Pharm. Bioprocess. 2, 491-498 (2014) doi: 10.4155/PBP.14.27.
14. Wen, J., Arthur, K., Chemmalil, L., Muzammil, S., Gabrielson, J. e Jiang, Y., J. Pharm. Sci. 101,955-964 (2012) doi: 10.1002/jps.22820.
15. Gabrielson, J.P. e Weiss, W.F., J. Pharm. Sci. 104, 1240-1245 (2015) doi: 10.1002/jps24393.
16. Cooney, B., Jones, S.D. e Levine, H., BioProcess Int. 14(6), 28-35 (2016). http://www.bioprocessintl.com/analytical/upstream-development/quality-by-design-for-monoclonal-antibodies-part-1-establishing-the-foundations-for-processdevelopment/
17. Cooney, B., Jones, S.D. e Levine, H., BioProcess Int. 14(8), 24-33 (2016). http://www.bioprocessintl.com/2016/quality-design-monoclonal-antibodies-part-2-process-design-space-control-strategies/
18. http://www.who.int/biologicals/biotherapeutics/rDNA_DB_final_19_Nov_2013.pdf
19 http://www.ema.europa.eu/docs/en_GB/document_library/Scientific_guideline/2009/09/WC500003935.pdf
20. http://www.fda.gov/downloads/Drugs/GuidanceComplianceRegulatoryInformation/Guidances/UCM496611.pdf
21. Jiang, Y. e Nahri, L.O., J. Am. Pharm. Rev. 9, 34-43 (2006).
22. Arthur, K.K., Dinh, N. e Gabrielson, J. P., J. Pharm. Sci. 104, 1544-1554 (2015) doi: 10.1002/jps.24313.
23. Shahrokh, Z., Salamat-Miller, N. e Thomas, J.J., Biophysical Analyses Suitable for Chemistry, Manufacturing, and Control Sections of the Biologic License Application (BLA), in: Biophysical Methods for Biotherapeutics: Discovery and Development Applications, T.K. Das (ed.) John Wiley & Sons, Hoboken NJ EUA páginas 317-353 (2014).
24. Lubiniecki, A., Volkin, D.B., Federici, M e outros, Biologicals 39, 9-22 (2011). doi: 10.1016/j.biologicals.2010.08.004.
25.http://www.fda.gov/Drugs/DevelopmentApprovalProcess/HowDrugsareDevelopedandApproved/ApprovalApplications/TherapeuticBiologicApplications/Biosimilars/
26.http://www.ema.europa.eu/docs/en_GB/document_library/Scientific_guideline/2015/01/WC500180219.pdf
27.http://www.who.int/biologicals/areas/biological_therapeutics/BIOTHERAPEUTICS_FOR_WEB_22APRIL2010.pdf
28 Bas, T.G. e Oliu Castillo, C., Biomed Res Int. 2016, 5910403 (2016). doi: 10.1155/2016/5910403.
29. Tsuruta, L.R., Lopes dos Santos, M. e Moro, A.M., Biotechnol Prog. 31, 1139-1149 (2015). doi: 10.1002/btpr.2066.
30 Kálmán-Szekeres, Z., Olajos, M. e Ganzler, K., J. Pharm. Biomed. Anal. 69, 185-195 (2012). doi: 10.1016/j.jpba.2012.04.037.
31 Berkowitz, S.A., Engen, J.R., Mazzeo, J.R. e Jones, G.B., Nat. Rev. Drug Discov. 11, 527-540 (2012). doi: 10.1038/nrd3746.
32.http://www.fda.gov/downloads/AdvisoryCommittees/CommitteesMeetingMaterials/Drugs/ArthritisAdvisoryCommittee/UCM510493.pdf
33.http://www.fda.gov/downloads/AdvisoryCommittees/CommitteesMeetingMaterials/Drugs/ArthritisAdvisoryCommittee/UCM484860.pdf
34. Jung, S.K., Lee, K. H., Jeon, J. W. e outros, MAbs 6, 1163-1177 (2014). doi: 10.4161/mabs.32221.
35.http://www.fda.gov/downloads/AdvisoryCommittees/CommitteesMeetingMaterials/Drugs/ArthritisAdvisoryCommittee/UCM510293.pdf
36. Liu, J., Eris, T., Li, C., Cao, S. e Kuhns, S., BioDrugs 30, 321-338 (2016). doi: 10.1007/s40259-016-0184-3.
37. Sinha-Datta, U., Khan, S. e Wadgaonkar, D., Biosimilars 5, 83-91 (2015) doi: 10.2147/BS.S85537 https://www.dovepress.com/label-free-interaction-analysis-as-a-tool-to-demonstrate-biosimilarity-peer-reviewed-article-BS