Ce livre blanc vous expliquera les principes de la titration calorimétrique isotherme et vous fournira des informations détaillées et des conseils concernant une large gamme d'études allant d'interactions simples à des mécanismes de liaison complexes et aux équilibres connexes.
La titration calorimétrique isotherme (ITC) est une technique d'analyse considérée comme une référence absolue dans l'étude des interactions intermoléculaires. Comme son nom l'indique, il s'agit d'une technique titrimétrique, c'est-à-dire d'une méthode volumétrique en laboratoire, utilisée dans l'analyse chimique quantitative (habituellement pour déterminer la concentration inconnue d'une substance non identifiée). Elle consiste à provoquer une réaction entre une solution de réactif, le titrant, et une solution de l'échantillon ou solution étalon. L'instrument ITC mesure la chaleur produite ou consommée par la réaction d'interaction lors de la titration pour déterminer le point final (lié à la stœchiométrie de l'interaction). En effectuant la titration à température et pression constantes, on peut obtenir lors d'une seule expérience ITC des informations concernant la constante d'association à l'équilibre, l'enthalpie de liaison et la stœchiométrie, ce qui permet de calculer facilement l'énergie de Gibbs et l'entropie de liaison. Ainsi, une expérience ITC unique permet d'accéder directement aux potentiels thermodynamiques principaux associés au processus d'interaction : énergie de Gibbs, enthalpie et entropie.
La haute sensibilité des instruments ITC modernes permet d'étudier la formation de complexes non covalents dans lesquels les molécules interagissent par le biais d'une combinaison d'interactions faibles, notamment des liaisons hydrogène, interactions électrostatiques, interactions de van der Waals et interactions hydrophobes. Il est donc possible d'étudier l'interaction entre tous types de molécules, des ions aux polymères, en passant par les nanoparticules et les biomolécules. La majorité des applications ITC sont focalisées sur les interactions biomoléculaires, telles que la formation de complexes de liaison protéine-ligand, protéine-protéine, protéine-ADN, protéine-membrane ou ADN-ligand bien définis (1, 2).
L'ITC permet une description quantitative exhaustive d'une interaction donnée en proposant des informations utiles à différents niveaux : 1) présence éventuelle d'interactions entre deux molécules données ; 2) stœchiométrie de liaison ; 3) affinité ou force de liaison de la formation de complexe par la constante d'association/de dissociation et l'énergie de liaison de Gibbs ; et 4) répartition de l'énergie de liaison de Gibbs en composantes enthalpique et entropique. En effet, concernant les interactions biomoléculaires, une analyse minutieuse des résultats expérimentaux, ainsi qu'une série d'expériences soigneusement planifiées dans des conditions différentes, permettent d'obtenir des informations supplémentaires pour mieux caractériser les interactions et traiter les thèmes suivants : 1) changements de conformation potentiels accompagnant le processus de liaison ; 2) processus concomitants association/dissociation supplémentaires (par exemple, échange net de protons et d'autres ions, ainsi que de molécules d'eau) accompagnant et modulant le processus de liaison ; et 3) phénomène de coopération impliquant des interactions homotropes (même ligand) ou hétérotropes (autre ligand). L'ITC permet donc d'accéder directement à des informations concernant l'interaction, ainsi que la régulation et la modulation des interactions biomoléculaires.
Une molécule donnée (habituellement dite « macromolécule », M) présente différents sites de liaison pour une autre molécule donnée (habituellement désignée « ligand », L). Cette distinction ne repose sur aucune supposition concernant la nature et la taille de ces molécules en interaction. Nous allons ici passer en revue les fondamentaux thermodynamiques des expériences ITC, ainsi que leur application, en privilégiant l'expression la plus simple de ce système (une macromolécule avec un seul site de liaison pour ligand, c'est-à-dire la stœchiométrie en 1:1), afin d'obtenir le plus d'informations possible. Une large majorité de cas d'études évoqués dans la littérature scientifique correspondent à ce système élémentaire. Cependant, la bibliographie comporte une description de systèmes plus complexes (interaction autre que 1:1) (3). En outre, vous trouverez des informations spécifiques concernant les instruments et protocoles expérimentaux dans les ouvrages de référence (4, 5).
L'ITC respecte les impératifs standard régissant une technique expérimentale destinée à l'étude des interactions de liaison, à savoir : 1) la propriété observable doit présenter des valeurs différentes dans différents états de liaison ; et 2) le signal mesuré doit être proportionnel à l'avancée du processus. L'ITC mesure directement la chaleur produite par un processus de réaction à pression constante, QT, proportionnelle à la charge d'enthalpie molaire associée à ce processus, ΔH (en prenant l'état de non liaison comme référence), et la quantité du complexe macromolécule-ligand formé :
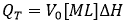 (1)
(1) |
où V0 correspond au volume de la cellule de calorimétrie et [M]T à la concentration totale de macromolécules dans la cellule. La première condition implique que l'enthalpie d'interaction (c'est-à-dire l'enthalpie de formation de complexe) ne doit pas être nulle. L'enthalpie d'interaction dépend de la température et d'autres variables expérimentales (telles que le pH, la force ionique ou la concentration du co-soluté). Il est donc relativement facile de trouver un ensemble de conditions expérimentales où cette valeur diffère de zéro. La deuxième condition est automatiquement satisfaite grâce à la première, puisque QT est proportionnelle à l'avancée du processus de liaison (indiqué comme une fraction de saturation de la macromolécule ou fraction molaire du complexe) :
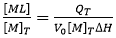 (2)
(2) |
Les Équations 1 et 2 peuvent être utilisées sans modèle afin d'estimer l'enthalpie de liaison, dans les conditions suivantes : 1) saturation presque complète de la macromolécule ([ML]/[M]T proche de 1) ; 2) soustraction adéquate des chaleurs de bruit de fond ; et 3) concentration en protéines déterminée selon un degré raisonnable de précision.
Par rapport à d'autres techniques biophysique d'étude des interactions de liaison, les principaux avantages de l'ITC sont les suivants : 1) caractérisation thermodynamique complète (stœchiométrie, constante d'association et enthalpie de liaison) par une seule expérience ; 2) la chaleur constitue un signal universel et ne nécessite donc pas de marqueur rapporteur (par exemple de chromophores ou fluorophores) ; 3) détermination directe à l'enthalpie de liaison (aucun besoin de suppositions supplémentaires concernant la relation de van't Hoff) ; 4) technique non-destructive ; 5) interaction en solution (aucun besoin d'immobilisation des réactifs) ; 6) possibilité d'effectuer des expériences avec des solutions présentant une densité optique ou des systèmes inhabituels (par exemple dispersions, organelles ou cellules intactes) ; et 7) rapidité relative (de 0,25 h/essai en expérience par injection unique à 2 h/essai en expérience par titration conventionnelle).
Bien entendu, l'ITC présente certains inconvénients, notamment : 1) le signal est proportionnel à l'enthalpie de liaison et les complexes non covalents peuvent présenter des enthalpies de liaison modestes (normalement, │ΔH│ < 40 kcal/mol, mais dans la majorité des cas │ΔH│ < 25 kcal/mol) ; 2) la chaleur est un signal universel et chaque processus contribue à la mesure globale de la chaleur, ce qui complique le calcul de la contribution émanant de la liaison ; 3) grande quantité d'échantillon requis, bien que cette valeur ait été considérablement réduite (exemple :~ 0,04 – 1 mg/essai pour une protéine de 50 kDa) ; 4) la technique est lente à bas débit (0,25 – 2 h/essai), ce qui fait d'elle une méthode non compatible avec une analyse à haut débit ; 5) les processus lents sur le plan cinétique peuvent ne pas être détectés ; 6) gamme limitée pour des affinités de liaison mesurées de manière fiable (constantes d'association de 104 à 109 M-1 pour expériences de titration conventionnelle) ; et 7) jusqu'à récemment, aucune donnée cinétique concernant les interactions de liaison n'était accessible.
Nombre de ces inconvénients ne sont pas limités à l'ITC, puisqu'on les retrouve dans bien d'autres techniques de liaison. Par ailleurs, certains peuvent être surmontés ou minimisés. Par exemple, les tampons présentant une enthalpie d'ionisation basse peuvent servir à réduire les effets de (dé)protonation non spécifiques et dépendant du tampon, qui contribuent à l'enthalpie mesurée. Il convient d'adopter la plus minutieuse des approches lors de la dissolution/solubilisation de réactifs, afin de garantir une correspondance de la composition entre toutes les solutions, et réduire la chaleur par injection en bruit de fond (également dite « chaleur de dilution »). En outre, la gamme pratique pour la détermination d'affinités peut être étendue par le biais de titrations de déplacement. De nouvelles méthodologies fiables d'estimation des données cinétiques concernant la liaison ont récemment été développées (6, 7).
Quand une macromolécule présentant un seul site de liaison et un ligand sont mélangés à certaines concentrations expérimentales, l'envergure de la réaction de liaison réversible, à savoir la concentration du complexe macromolécule-ligand, [ML], est régie par la constante d'association à l'équilibre, Ka, que l'on obtient comme suit :
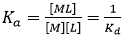 (3)
(3) |
où Kd est la constante de dissociation à l'équilibre pour le complexe, [M] et [L] sont les concentrations de macromolécule et ligand libres. Les deux constantes d'équilibre sont liées à la loi (standard) de Gibbs sur l'énergie de liaison, ΔG :
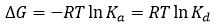 (4)
(4) |
où R correspond à la constante des gaz et T la température thermodynamique ou absolue. Plus la constante d'association est grande, plus la constante de dissociation est modeste et plus l'énergie de liaison de Gibbs devient négative, donnant lieu à un complexe plus fort.
À partir de l'Éq. 3, il est évident que si [L] = 1/Ka = Kd, la macromolécule est distribuée en parts égales parmi les états libre et liée. L'Éq. 3 permet d'obtenir la relation hyperbolique bien connue entre la fraction molaire du complexe macromolécule-ligand et la concentration de ligand libre :
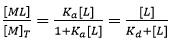 (5)
(5) |
permettant de calculer facilement la concentration du complexe macromolécule-ligand formé et, donc, la chaleur associée avec cette formation, en employant une multiplication par la concentration totale de protéines et l'enthalpie de liaison molaire. Malheureusement, dans une expérience par titration, la concentration de ligands libres est inconnue et les concentrations contrôlées sont celles de la concentration totale de macromolécules et de ligands. Par conséquent, il est impossible d'utiliser l'Éq. 5 dans le cadre de l'analyse de la titration.
L'énergie de liaison de Gibbs peut être répartie entre ses contributions enthalpique, ΔH, et entropique, -TΔS :
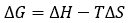 (6)
(6) |
Le profil thermodynamique de liaison de base pour une interaction donnée comprend les trois principaux paramètres de liaison, énergie de Gibbs (calculée à partir de la constante Ka d'association à l'équilibre), enthalpie et entropie de liaison. L'association de ces trois potentiels thermodynamiques permet d'obtenir des informations utiles et fondamentales sur le processus de liaison (par exemple le type d'interaction intermoléculaire dynamisant l'interaction).
Malgré la relation entre les potentiels thermodynamiques, l'affinité de liaison (sous forme de constantes d'équilibres ou d'énergie Gibbs de liaison) n'est pas liée à l'enthalpie ou l'entropie de liaison. En d'autres termes, une affinité élevée ou basse n'est pas forcément associée à une certaine distribution d'affinité dans les contributions enthalpiques et entropiques. Par conséquent, deux ligands présentant une affinité similaire peuvent démontrer des profils de liaison thermodynamique très différents, voyant l'un d'eux afficher une liaison à dominance enthalpique (la contribution enthalpique est négative, favorable à la liaison ; la contribution entropique est positive et défavorable à la liaison), tandis que l'autre ligand affiche une liaison à dominance entropique (la contribution enthalpique est positive, défavorable à la liaison ; la contribution entropique est négative, favorable à la liaison).
Il convient de préciser que le profil thermodynamique de base dépend de la température et d'autres variables environnementales (telles que le pH et la force ionique). Par conséquent, il importe d'évaluer ce type de dépendance afin de prédire le comportement du complexe macromolécule-ligand dans d'autres environnements (par exemple, un complexe physiologique protéine-protéine soumis à une acidification de son environnement sur le plan intracellulaire). Les modifications structurelles survenant chez les partenaires de liaison (telles que des mutations dans la protéine de liaison, des dérivées fonctionnelles du ligand) entraîneront également une modification du profil thermodynamique de liaison.
Le principal objectif d'une expérience de titration consiste à déterminer la constante d'association à l'équilibre, l'enthalpie de liaison et la stœchiométrie, et, par extension, le profil thermodynamique, ainsi que sa dépendance envers des variables environnementales et des changements structurels chez les partenaires de liaison (dans la mesure du possible.
Comme nous l'avons vu plus tôt, une expérience d'ITC permet d'obtenir l'intégralité du profil thermodynamique de base pour une interaction de liaison donnée. La méthodologie expérimentale consiste à effectuer une série d'injections de titrant (habituellement le ligand) à l'aide d'une seringue dans la solution étalon (habituellement la macromolécule) au sein de la cellule selon un processus entièrement automatisé, tout en maintenant le système dans des conditions isobares quasiment isothermes. Grâce à l'injection séquentielle du ligand, l'équilibre chimique est perturbé par les changements progressifs de la concentration du réactif provoqués par les injections de ligand. Le système passe par une suite d'états d'équilibre de compositions différentes, puisque la répartition entre la macromolécule libre et la macromolécule liée au ligand dépend de la constante d'équilibre (d'association ou de dissociation) et des concentrations totales de macromolécules et de ligands :
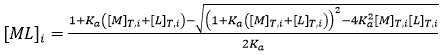 (7)
(7) |
où l'indice i indique le nombre d'injection au fil de la titration, et les concentrations totales de macromolécules et de ligands après chaque injection s'obtiennent par :
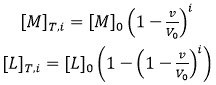 (8)
(8) |
où [M]0 et [L]0 correspondent respectivement à la concentration initiale de macromolécule dans la cellule et la concentration de ligand dans la seringue, v est le volume d'injection et le facteur (1-v/V0) exprime la dilution qui a lieu dans une cellule calorimétrique fonctionnant à volume constant. Chaque transition entre deux états successifs, provoquée par l'injection de ligand, s'accompagne de la libération ou l'absorption de chaleur (à pression constante, liée au changement global d'enthalpie), proportionnellement au changement de concentration de complexe dû à cette injection et à l'enthalpie molaire :
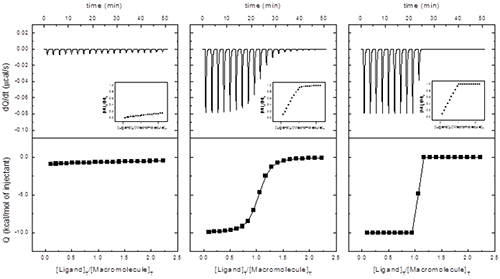
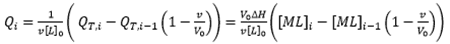 (9)
(9) |
qui démontre clairement que l'ITC constitue une technique progressive ou à différences finies. La chaleur associée à une injection donnée dans l'Éq. 9 a été normalisée par le nombre de moles de ligand injectées, divisé par v[L]0. La Figure 1 illustre une titration calorimétrique typique pour une macromolécule possédant un seul site de liaison pour ligand. Une analyse par régression non linéaire des données expérimentales en utilisant l'Éq. 9 permet d'estimer la constante d'association à l'équilibre, l'enthalpie de liaison et la stœchiométrie, et de calculer ensuite facilement l'énergie de Gibbs et l'entropie de liaison. Des modèles simples, comme celui qui est considéré ici, peuvent être utilisés avec des procédures d'ajustement fournies par le fabricant de l'instrument.
|
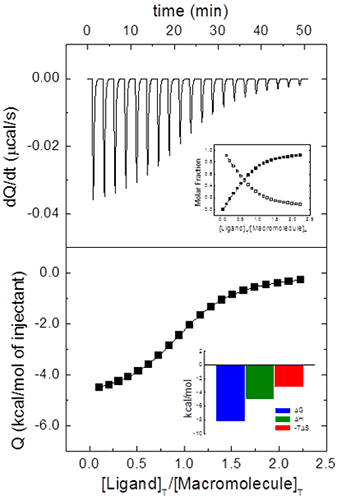
Même pour ce système simple comportant un site de liaison unique pour ligand, il est courant d'inclure un paramètre supplémentaire N pour multiplier [M]0 dans l'Éq. 8 ou [M]T,i dans l'Éq. 7. Ce paramètre représentera soit la stœchiométrie (dans un modèle pour une macromolécule comportant N sites de liaison identiques et indépendants (modèle dit d'« un ensemble de sites de liaison ») soit la fraction de macromolécules actives (aptes à la fixation) avec un seul site de liaison de ligand. Bien souvent, les titrations ITC constituent la meilleure façon d'estimer la concentration d'une macromolécule (active), car les méthodes standard (telles que l'absorption d'UV, les méthodes calorimétriques ou l'analyse élémentaire) pâtissent d'erreurs systématiques ou fournissent une concentration totale de protéines, sans offrir de distinction entre une macromolécule fonctionnelle et non fonctionnelle (protéine mal repliée, par exemple). Les valeurs fractionnaires de N pourraient donc être liées aux incertitudes concernant les concentrations des deux réactifs. D'ordinaire, il est possible de déterminer avec précision la concentration en ligands à condition de disposer de données spectroscopiques et/ou de procédures de pesée précises, non sujettes à une dégradation rapide. Cependant, même en faisant preuve d'une extrême minutie, il n'est pas rare d'obtenir une fraction non-négligeable de protéine dans une conformation compétente non liante. Il est donc possible de normaliser la concentration de protéines nominale à l'aide du paramètre N afin d'obtenir une valeur N non fractionnelle (Figure 2), permettant d'effectuer la « titration » de la solution de protéines (c'est-à-dire de déterminer sa concentration exacte à partir de la concentration connue d'un réactif). Par ailleurs, dans la mesure où l'estimation de l'enthalpie de liaison dépend de la concentration en ligands dans la seringue, alors que le facteur de normalisation N dépend des deux concentrations, en principe, en connaissant la valeur prévue de l'enthalpie de liaison, il est possible d'estimer la concentration des deux réactifs à partir d'une titration : la concentration en ligands est estimée à l'aide de l'enthalpie de liaison, et, par la suite, la concentration de macromolécules est estimée à l'aide de N. Lors de l'étude d'une protéine donnée, il s'avère très pratique de disposer d'un ligand d'étalonnage pour lequel les paramètres de liaison sont connus avec un degré de précision raisonnable, ce qui est utile pour le contrôle qualité dans les phases de production de protéines.
|
Pour appliquer l'Éq. 9 correctement dans le cadre de l'analyse d'une titration expérimentale et obtenir de bonnes estimations des paramètres de liaison, il est nécessaire de régler le problème de la « chaleur de dilution ». En plus de la chaleur associée à l'interaction macromolécule-ligand, l'injection de la solution de ligand dans la cellule peut s'accompagner d'une quantité de chaleur de bruit de fond non négligeable, potentiellement causée par la dilution du ligand, effet mécanique de l'injection (par exemple turbulence ou mélange), ou équilibrage de disparités mineures de composition entre la solution de titrant et la solution étalon (telles que des différences modestes de pH, ou de concentration de DMSO ou glycérol quand ces additifs sont requis). Il existe trois manières de supprimer la contribution de cet effet de chaleur non spécifique : 1) exécuter une expérience de contrôle en diluant le ligand dans le tampon et en soustrayant les chaleurs résultantes de la titration ligand-macromolécule ; 2) calculer la moyenne des chaleurs associées aux dernières injections (aux abords de la saturation de macromolécule) dans la titration ligand-macromolécule et soustraire cette valeur dans les données ; et 3) introduire dans l'Éq. 9 un paramètre ajustable supplémentaire, Qd, pour tenir compte de la chaleur de bruit de fond par injection. La troisième option est à privilégier, car l'expérience de dilution ne reflète pas correctement l'effet d'injection en bruit de fond quand la macromolécule est présente (la soustraction d'expérience de contrôle de la titration pourrait ne pas donner de résultats satisfaisants). Bien souvent, le seuil de saturation de macromolécule n'est pas entièrement atteint (la moyenne des chaleurs associées avec les derniers pics pourrait n'avoir aucune signification, comme l'illustre la Figure 1). Hormis cela, il est crucial d'effectuer régulièrement des expériences de contrôle (c'est-à-dire de diluer le ligand dans un tampon pour exclure des phénomènes inattendus, tels que l'auto-association des ligands), ainsi que des essais de calibration, selon des protocoles électriques ou, idéalement, chimiques (en utilisant des processus chimiques bien définis).
Les avancées en matière de miniaturisation et d'automatisation ont permis de créer des calorimètres hypersensibles, tels que le MicroCal™ Auto-iTC200, qui permet la mesure précise de la chaleur de réaction par injection de quelques microcalories, à partir d'une fraction de mg (des dizaines de microgrammes) de produit dans la cellule de calorimétrie. De par leur simplicité, leur fiabilité et leur précision, les méthodes de titration calorimétrique sont devenues une technique standard, largement plébiscitée dans le domaine des études d'interactions biophysiques.
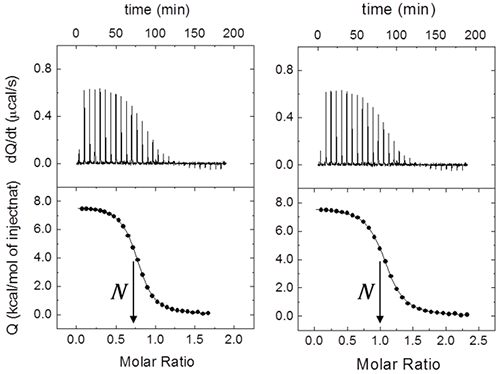
En théorie, il est possible d'estimer par le biais d'une titration unique la constante d'association à l'équilibre, l'enthalpie de liaison et la stœchiométrie. Cependant, la fiabilité de ces mesures dépend de l'affinité de liaison et de la configuration expérimentale. Il est possible de calculer un paramètre sans dimension c = Ka[M]0 = [M]0/Kd et : 1) si 1 < c < 10 000, les trois paramètres de liaison peuvent être estimés simultanément et précisément ; 2) si c > 10 000, seules l'enthalpie de liaison et la stœchiométrie peuvent être déterminées ; et 3) si c < 1, seule la constante d'association à l'équilibre peut être déterminée, puisque l'enthalpie de liaison et la stœchiométrie seront corrélées (cependant, si N peut être établi pendant l'analyse de régression et qu'on atteint un seuil suffisant de saturation macromoléculaire, l'enthalpie de liaison peut être déterminée avec fiabilité). La plage indiquée pour l'estimation fiable de l'affinité de liaison (1 < c < 10 000) est relativement généreuse. Bien que les valeurs de c légèrement supérieures à 1 000 ne représentent habituellement pas de problème pour l'estimation de l'affinité de liaison, la plage optimale est de 10 < c < 1 000. La Figure 3 illustre les trois scénarios selon la valeur de c. Il est intéressant de constater que le scénario idéal correspond à une titration effectuée à l'aide d'une concentration de macromolécules nettement supérieure à la constante de dissociation à l'équilibre Kd. Cependant, dans une titration spectroscopique standard (par exemple par fluorescence, résonance magnétique nucléaire ou dichroïsme circulaire), il est préférable d'utiliser une concentration de macromolécules avoisinant la valeur Kd pour obtenir des résultats optimaux.
|
Dans la mesure où la courbe de titration dépend du paramètre c, selon une affinité de liaison, il devrait être possible de moduler la courbe en sélectionnant la concentration de macromolécules. Bien que la situation idéale corresponde à une valeur 1 < c < 10 000, il n'est pas toujours possible d'opérer dans cette fenêtre, car nous risquons d'utiliser des concentrations de réactif très basses (si l'affinité de liaison est très élevée), ce qui peut entraîner un signal calorimétrique bas, ou, en utilisant des concentrations de réactifs d'une valeur excessivement élevée, entraîner un signal calorimétrique important (dépassant la gamme instrumentale) ou des concentrations dépassant les seuils pratiques.
Si notre système expérimental présente une affinité très élevée (c > 10 000) ou très basse (c < 1), les titrations de décalage tenant compte d'un ligand à affinité modeste permettent de surmonter ce problème (8).
Dans la mesure où l'enthalpie et l'entropie de liaison témoignent d'interactions d'un type très différent, le profil de liaison thermodynamique permet d'obtenir des informations très utiles sur les interactions interatomiques qui sous-tendent la liaison et sur le mode d'interaction entre la macromolécule et le ligand. La contribution enthalpique reflète globalement les interactions spécifiques entre les deux réactifs (formation de liaisons hydrogène, forces de van der Waals, interactions électrostatiques et hydrophobes), tandis que la contribution entropique reflète des interactions non spécifiques entre la macromolécule et le ligand (désolvation de l'interface de liaison, complémentarité géométrique entre le ligand et le site de liaison de la macromolécule et rigidité structurelle du ligand). Il est donc possible d'établir que les ligands à dominance enthalpique s'attachent à la macromolécule parce qu'ils exercent des interactions spécifiques avec la cible (c'est-à-dire qu'ils « aiment » se trouver sur le site de liaison), tandis que les ligands à dominance entropique s'attachent à la macromolécule parce qu'ils sont très hydrophobes et présentent une géométrie et une rigidité structurelle adéquate (c'est-à-dire qu'ils « n'aiment pas » se trouver dans l'eau).
Il est important de souligner que ces changements de potentiels thermodynamiques accompagnant la liaison reflètent non seulement la création d'un réseau considérable d'interactions intermoléculaires (c'est-à-dire des liaisons hydrogène, des forces van der Waals et des interactions électrostatique et hydrophobes) entre macromolécule et ligand, mais également la perturbation d'interactions similaires pour les deux molécules avec les molécules de solvant. Par conséquent, ces changements représentent le résultat net d'interactions contradictoires dans lesquels les molécules de solvant jouent un rôle important. Globalement, l'affinité de liaison est le résultat net de ces différentes contributions enthalpiques et entropiques.
En marge des informations utiles sur le mode de liaison entre la macromolécule et le ligand, la répartition d'énergie de Gibbs en contributions enthalpiques et entropiques présente des conséquences importantes. Il a été démontré que l'énergie de liaison de Gibbs est plutôt insensible aux fluctuations de variables d'environnement et aux modifications chez les partenaires de liaison. Cette observation est étroitement liée à la compensation enthalpie-entropie (c'est-à-dire que les changements de contribution enthalpique pour la liaison sont généralement associés à des changements opposés de contribution entropique, ce qui permet d'éviter quasiment toute modification de l'énergie de liaison de Gibbs) et aux difficultés d'amélioration de l'affinité de liaison du ligand en intervenant au niveau de la structure du ligand. Cependant, il est établi que les potentiels connexes liés à l'énergie de Gibbs en dérivation de température (c'est-à-dire l'enthalpie et l'entropie de liaison) sont plus sensibles aux variables d'environnement et aux modifications chez les partenaires de liaison, ce qui entraîne des paramètres de liaison plus favorables à une interprétation. Certaines données acquises suggèrent que la répartition de l'énergie de Gibbs dans ses contributions enthalpique et entropique présente des conséquences importantes concernant l'optimisation des affinités de liaison du ligand et la minimisation de la susceptibilité des ligands aux changements structurels (induits par des mutations ou causés par une variabilité naturelle) dans la cible macromoléculaire (9, 10).
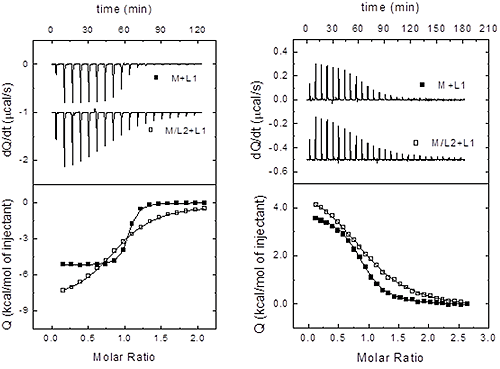
Une macromolécule donnée est susceptible d'entrer en interaction avec plus d'un ligand. Dans l'exemple particulier d'une macromolécule interagissant avec deux ligands différent, hormis la caractérisation des interactions de liaison avec la macromolécule pour chaque ligand, quand aucune autre information structurelle n'est disponible, il serait intéressant d'évaluer la capacité éventuelle des ligands à former des liaisons compétitives (directement par le biais d'une liaison au même site de liaison, ou indirectement par le biais d'un changement de conformation empêchant la liaison du deuxième ligand). L'ITC s'avère particulièrement utile pour cette évaluation car, contrairement à d'autres techniques employant un seul paramètre de liaison (affinité de liaison) pour établir des distinctions entre des liaisons indépendante, coopératives ou compétitives, l'ITC permet d'utiliser deux paramètres de liaison (affinité de liaison et enthalpie) pour établir des distinctions entre les trois cas de figure.
La configuration de cette procédure expérimentale est assez simple : 1) effectuer deux titrations en injectant chaque ligand dans la macromolécule ; et 2) effectuer une troisième titration en injectant les ligands dans la macromolécule pré-liée au deuxième ligand (deuxième ligand en excès, si possible). Toutes les titrations, qu'elles soient binaires ou ternaires, peuvent être analysées à l'aide du modèle de site de liaison unique (si le deuxième ligand est en excès concernant la concentration de macromolécules, la titration ternaire peut être analysée en tant que titration binaire). Il est alors possible d'obtenir trois ensembles de paramètres de liaison (constante d'équilibre et enthalpie) à partir des trois titrations : Ka,1 et ΔH1 pour l'interaction du ligand 1, Ka,2 et ΔH2 pour l'interaction du ligand 2, et Ka,12 et ΔH12 pour l'interaction du ligand 1 en présence du ligand 2. La différenciation entre ces différents scénarios repose sur une comparaison de ces trois ensembles de paramètres. Comme nous allons le voir, dans la mesure où les liaisons indépendantes et compétitives représentent des cas particuliers de liaisons coopératives (la liaison compétitive constitue notamment le cas limite pour la coopération négative maximale), nous aborderons l'exemple général de coopération, puis étudierons les cas particuliers de liaisons compétitives et indépendantes.
Si les deux ligands établissent une liaison coopérative, les paramètres de liaison apparente pour le ligand 1 en présence du ligand 2 (pour une certaine concentration) sont les suivants :
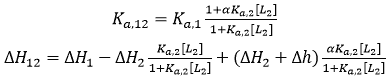 (14)
(14) |
où [L2] correspond à la concentration libre du ligand 2 dans la cellule calorimétrique (si le ligand 2 est utilisé à une concentration totale nettement supérieure à celle de la macromolécule, il est possible d'atteindre une approximation de sa concentration libre par sa concentration totale), α est la constante d'interaction de coopération (0 < α < 1 correspond à une coopération négative, tandis que 1 < α < +∞ correspond à une coopération positive), et Δh est l'enthalpie de liaison de coopération. L'Éq. 14 fournit les paramètres de liaison apparente du ligand 1 en interaction avec la macromolécule en présence du ligand 2 à une concentration donnée. Cependant, les paramètres de liaison intrinsèques pour l'interaction du ligand 1 avec la macromolécule pré-liée au ligand 2 sont différents : si le ligand 2 est lié à la macromolécule, le ligand 1 interagit avec la macromolécule comportant une constant d'association à l'équilibre intrinsèque αKa,1, et une enthalpie de liaison intrinsèque ΔH1 + Δh (c'est-à-dire les concentrations de saturation du ligand 2 dans l'Éq. 14).
Si les deux ligands se lient indépendamment, la liaison du ligand 2 à la macromolécule dans la titration ternaire n'affectera pas le déroulement de la titration, puis, α = 1 et Δh = 0 :
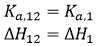 (15)
(15) |
Si les deux ligands se lient de manière compétitive, la liaison du ligand 2 à la macromolécule dans la titration ternaire bloquera la liaison du ligand 1, puis α = 0:
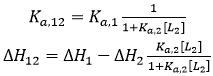 (16)
(16) |
Par conséquent, une fois que les trois ensembles de paramètres de liaison sont obtenus par analyse des titrations binaire et ternaire, nous devons appliquer de manière consécutive les équations précédentes en tant que critères de différenciation, en commençant par le test d'indépendance (Éq. 15), suivi du test de compétition (Éq. 16). Si aucun de ces tests n'est effectué, en tenant compte des incertitudes expérimentales dans les paramètres de liaison estimés, les deux ligands établissent une liaison coopérative (c'est-à-dire ni indépendante ni compétitive). La Figure 4 illustre cette procédure dans deux systèmes expérimentaux.
|
Nous avons vu précédemment que les contributions enthalpiques et entropiques fournissent des informations fondamentales sur l'interaction de liaison. Étant donné que la contribution entropique est calculée à partir de l'enthalpie de liaison estimée et de l'énergie de Gibbs, il est extrêmement important d'obtenir des estimations fiables de ces paramètres de liaison et d'éliminer les contributions extrinsèques potentielles.
Une contribution potentielle à l'énergie de Gibbs et à l'enthalpie de liaison provient de l'échange de protons lors de la liaison entre le complexe macromolécule-ligand et la solution en vrac. Si certains groupes ionisables (issus de l'un des partenaires de liaison) subissent un changement de pKa en raison d'un changement d'exposition au solvant suite à la liaison du ligand (par liaison directe ou induite par le changement de conformation), on constatera un changement de saturation de fraction de protons entre chaque groupe ionisable dans les réactifs libres et dans le complexe. Ce phénomène entraîne un échange net de protons (acquisition ou libération), compensé par le tampon afin de préserver un pH constant, ce qui se traduit par une contribution de l'énergie de Gibbs et de l'enthalpie d'ionisation du tampon aux paramètres généraux de liaison. Heureusement, si le tampon sélectionné présente une valeur pKa similaire au pH expérimental, l'énergie de liaison de Gibbs mesurée ne présentera pas de contribution significative venant du processus d'ionisation du tampon. Au contraire, si le tampon sélectionné présente une enthalpie d'ionisation considérable, ΔHB,ion, l'enthalpie de liaison mesurée, ΔH, peut contenir une contribution significative du processus d'ionisation de tampon :
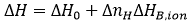 (12)
(12) |
où ΔH0 est l'enthalpie de liaison indépendante du tampon, et ΔnH représente le nombre net de protons échangés entre le complexe et la solution en vrac dans le cadre de la liaison de ligand. La valeur du paramètre ΔnH pourrait devenir un nombre non entier, car il s'agit d'un paramètre statistique servant à tenir compte de la différence dans la saturation globale de fraction de protons entre les réactifs libres et le complexe. Il indique une protonation nette du complexe quand il est positif et une déprotonation nette du complexe quand il est négatif. Il est clair que la contribution du tampon dans l'Éq. 12 pourrait dominer l'enthalpie globale de liaison, ce qui ne permettra pas toujours de déterminer l'enthalpie de liaison du ligand, mais peut s'avérer utile pour amplifier le signal calorimétrique quand l'enthalpie de liaison du ligand est faible. D'après l'Éq. 12, en exécutant les expériences ITC dans les mêmes conditions (température, pH, force ionique, etc.) et en utilisant des tampons présentant différentes enthalpies d'ionisation, il est possible d'évaluer les processus d'échange de protons associés à la liaison de ligands et d'estimer l'enthalpie de liaison indépendante du tampon (pour établir de manière fiable le profil de liaison thermodynamique sans contributions extrinsèques à la liaison) et le nombre net de protons échangés. La Figure 5 illustre cette procédure expérimentale. Il est également possible d'utiliser des tampons à enthalpie d'ionisation très basse ou négligeable (tels que le phosphate ou l'acétate) et de déterminer l'enthalpie de liaison indépendante du tampon dans une seule expérience, sans devoir utiliser l'Éq. 12. Le corolaire de cette observation est qu'il n'existe pas de mauvais tampon en calorimétrie : un tampon peut déformer et considérablement moduler l'enthalpie de liaison observée (si les processus d'échange de protons se déroulent lors de la liaison et que l'enthalpie d'ionisation du tampon est suffisamment importante), mais cela peut contribuer à détecter l'interaction en amplifiant le signal de chaleur. Il existe une procédure permettant de supprimer sa contribution à l'enthalpie mesurée.
|
Il est intéressant de souligner que la valeur de ΔnH permet de prédire le comportement de l'affinité de liaison suite à un changement de pH. Si la liaison de ligand s'accompagne de processus d'échange de protons, le profil de liaison thermodynamique sera dépendant du pH. Ainsi, en vertu d'une relation chimique bien connue, la constante d'association à l'équilibre et l'énergie de liaison de Gibbs seront dépendants du pH :
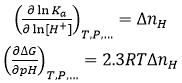 (13)
(13) |
En connaissant ΔnH grâce aux expériences effectuées à pH constant et en utilisant l'Éq. 12, nous pouvons prédire quantitativement le changement d'affinité de liaison pour les changements pH finis (dans une gamme de variation de pH, à savoir |ΔpH| ≤ 2) ; remarquons notamment que si ΔnH > 0 (protonation nette du complexe), l'affinité de liaison baissera en fonction du pH, tandis que si ΔnH < 0 (déprotonation nette du complexe), l'affinité de liaison augmentera en fonction du pH.
La capacité calorifique de liaison (à pression constante) est la dérivée de la température de l'enthalpie de liaison :
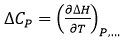 (10)
(10) |
facilement accessible sur le plan expérimental en effectuant des titrations à différentes températures et en représentant l'enthalpie de liaison expérimentale observée sous forme de fonction de la température. Sur une plage modeste de températures (par exemple de 10 à 40°C), l'enthalpie de liaison observée présente souvent une tendance linéaire, permettant d'estimer la capacité calorifique de liaison sous forme de pente de la régression linéaire.
La capacité calorifique de liaison est fortement liée à l'enfouissement et à la désolvation des interfaces de liaison. La réduction de surface accessible sur la macromolécule et le ligand, avec des contributions de déshydratation des régions polaires et apolaires, entraîne une réduction de la capacité calorifique du système (capacité à emmagasiner et échanger de l'énergie calorifique) : la déshydratation des régions polaires rend la capacité calorifique plus positive, tandis que la déshydratation des régions apolaires rend la capacité calorifique plus négative, cette dernière se montrant cependant prédominante dans les interfaces de liaison. D'autres phénomènes (par exemple une réduction de la mobilité locale et des degrés de vibration libre dans la macromolécule et le ligand) contribuent également à la capacité calorifique de liaison, mais à un degré moindre (<10 %). Le lien entre la capacité calorifique de liaison et la réduction de surface accessible (polaire et apolaire) lors de la formation de complexe permet d'établir des relations linéaires entre les deux quantités. La première est accessible via ITC et la deuxième par des techniques structurelles (telles que la diffraction par rayons X ou la résonance magnétique nucléaire).
La réussite de cette paramétrisation structurelle de la capacité calorifique de liaison permet d'évaluer des changements potentiels de conformation associés à la liaison du ligand à partir des informations structurelles du complexe, même quand les structures des partenaires de liaison libres ne sont pas disponibles. En supposant une formation de complexe rigide ne présentant pas de changement de conformation, les structures des partenaires de liaison libres peuvent être obtenues directement à partir de la structure du complexe et on peut calculer les changements de surface accessible (polaire et apolaire). À partir de ces changements, il est possible d'estimer la capacité calorifique de liaison correspondant à l'interaction de liaison à la structure rigide. La déviation de la capacité calorifique de liaison par rapport à la capacité calorifique de liaison déterminée de manière expérimentale est directement liée à l'envergure du changement potentiel de conformation accompagnant la liaison de ligand.
Comme nous l'avons expliqué ci-dessus, l'enthalpie de liaison observée adopte habituellement un comportement linéaire sur une plage de températures limitée. Dans certains cas, cette observation n'est pas valide, et une tendance incurvée avec pente variable apparaît (la pente devient progressivement plus négative à mesure qu'augmente la température), ce qui implique une capacité calorifique de liaison dépendante de la température. Ce phénomène se produit quand le changement de conformation accompagnant la liaison est associé à un équilibre de conformation dépendant de la température au sein de la plage de températures opérationnelles : l'explication la plus simple est que l'un des partenaires de liaison présente un équilibre entre deux conformations, native et partiellement dépliée, et que l'augmentation de température repousse progressivement l'équilibre vers la conformation non-native. On observe alors une transition progressive d'une capacité calorifique de liaison modeste (liaison du ligand à la conformation native) vers une grande capacité (liaison du ligand à la conformation non-native partiellement dépliée). Globalement, un changement de conformation induit par la température s'oppose à un changement de conformation induit par le ligand. Le changement de conformation dépendant de la température explique la courbe présente sur le tracé d'enthalpie.
Quelques remarques corolaires s'imposent concernant les changements de conformation associés aux liaisons de ligands. Si aucun changement de conformation ne se produit suite à la liaison de ligand, une tendance linéaire peut s'observer sur le tracé de l'enthalpie de liaison comparée à la température. Cependant, cette condition ne suffit pas. En effet, une tendance linéaire sur ce tracé ne constitue pas une indication de l'absence de changements de conformation associés aux liaisons de ligands. Par conséquent, une tendance linéaire dans l'enthalpie de liaison indique que le changement de conformation éventuel se produit sur la même envergure le long de la plage de températures (approximativement le même changement de populations des états de conformation, quelle que soit la température. En d'autres termes, il n'existe pas d'équilibre de conformation dépendant de la température). Si le changement de conformation se produit à une envergure différente selon un schéma dépendant de la température au sein de la plage de températures expérimentale (changement différent de populations des états de conformation, selon la température. En d'autres termes, il existe un équilibre de conformation dépendant de la température, modulé par la liaison de ligands). On observera alors un tracé incurvé avec une pente variable dans l'enthalpie de liaison mesurée.
Par ailleurs, la capacité calorifique de liaison permet une extrapolation des potentiels thermodynamiques (énergie de Gibbs, enthalpie et entropie de liaison) obtenus à une certaine température de référence T0 sur une plage de températures donnée, afin d'évaluer le comportement de l'affinité de liaison avec changement de température :
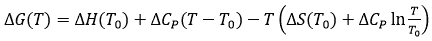 (11)
(11) |
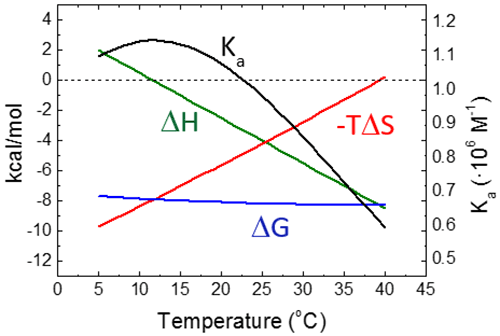
La Figure 6 illustre la dépendance à la température du profil de liaison thermodynamique pour une interaction de liaison donnée, en supposant une capacité calorifique de liaison constante.
|
Les avancées dans l'instrumentation ITC (automatisation, miniaturisation) ont permis la création d'un modèle d'ITC MicroCal Auto-iTC200, automatisé et à haut débit, présentant un profil de faible consommation d'échantillons/durée par essai expérimental (~50 essais/jour). De nouveaux développements méthodologiques, tels que la méthode d'injection unique, pourraient considérablement augmenter le débit, tout en préservant la précision de la méthode (11).
L'ITC est une technique efficace pour la caractérisation des interactions, notamment les interactions de biomolécules dans une large plage d'affinités de liaison (des affinités faibles observées dans les mécanismes physiologiques aux affinités très élevées observées dans les phases de conception et de développement de médicaments).
L'ITC peut jouer un rôle clé dans de nombreux secteurs, notamment : 1) fonction et régulation des protéines, caractérisation du mode d'interaction, spécificité et interactions allostériques (par exemple mécanismes d'enzymes, transmission de signaux aux récepteurs) ; 2) synthèse de protéines et modification de leur fonction ; 3) caractérisation de cibles pharmacologiques/biotechnologiques ; 4) sélection de pistes dans la découverte de médicaments ; 5) optimisation de pistes dans le développement de médicaments, selon une relation quantitative-structure-activité énergétique ; 6) développement de vecteurs pharmaceutiques pour améliorer l'administration thérapeutique ; 7) essais pré-cliniques (par exemple liaison plasma protéine, effets secondaires potentiels par cibles non souhaitables) ; 8) banc d'essai expérimental pour développement et perfectionnement des modèles computationnels relatifs aux interactions de protéines (ancrage, analyse virtuelle, prédiction des paramètres énergétiques d'interaction…); et 9) contrôle qualité pour la production de protéines/produits biologiques dans le secteur pharmaceutique, les produits de beauté, l'alimentation, le nettoyage et la biotechnologie.
Ce Livre blanc a été rédigé par le Dr. Adrian Velazquez-Campoy, travaillant actuellement en tant que chercheur dans la Fondation ARAID, auprès de l'Institut de bio-informatique et de Physique des Systèmes Complexes (Université de Saragosse), Mariano Esquillor s/n, 50018, Saragosse, Espagne. email : adrianvc@unizar.es
Falconer, R.J. et Collins, B.M. Survey of the year 2009: Applications of isothermal titration calorimetry. J. Mol. Recognit. 24, 1-16, (2011).
Ghai, R. et al. Applications of isothermal titration calorimetry in pure and applied research - Survey of the literature from 2010. J. Mol. Recognit. 25, 32-52 (2012).
Vega, S. et al. A unified framework based on the binding polynomial for characterizing biological systems by isothermal titration calorimetry. Methods, in press (2015).
Doyle, M.L. Titration microcalorimetry. Curr. Protoc. Prot. Sci. Unit 20.4 (2001).
Lewis, E.A. et Murphy, K.P. Isothermal titration calorimetry. Methods Mol. Biol. 305, 1-15 (2005).
Sigurskjold, B.W. Exact analysis of competition ligand binding by displacement. Isothermal titration calorimetry. l Biochem. 277, 260-266 (2000).
Burnouf, D. et al. kinITC: A new method for obtaining joint thermodynamic and kinetic data by isothermal titration calorimetry. J. Am. Chem. Soc. 134:559-565 (2012).
Velazquez-Campoy, A. et al. Isothermal titration calorimetry. Curr. Protoc. Cell Biol. Unit 17.8 (2004).
Freire, E. A thermodynamic approach to the affinity optimization of drug candidates. Chem. Biol. Drug Des. 74, 468-472 (2009).
Freire, E. Isothermal titration calorimetry: Controlling binding forces in lead optimization. Drug. Discov. Today Technol. 1, 295-299 (2004).
Markova, N. et Hallén, D. The development of a continuous isothermal titration calorimetric method for equilibrium studies. Anal. Biochem. 331, 77-88 (2004).