La estabilidad de una proteína biofarmacéutica es fundamental para el éxito o el fracaso del desarrollo de un medicamento viable. La estabilidad de las proteínas es importante para la producción, la fabricación, la formulación, el almacenamiento a largo plazo, la administración al paciente y la eficacia. Las proteínas con una mayor estabilidad probablemente presentarán menos problemas durante el proceso de fabricación, son más rentables de producir y tienen más probabilidades de mantenerse funcionales durante la formulación y el almacenamiento, sin alteración química o agregación. En el enfoque de "calidad basada en el diseño" (QbD, por sus siglas en inglés) para el desarrollo biofarmacéutico, la caracterización de la estabilidad es parte de una evaluación de la "capacidad de desarrollo" y la "capacidad de convertirse en fármaco" de una molécula candidata, así como durante el desarrollo del proceso y la fabricación. Los datos de estabilidad también se incorporan en la caracterización e identificación de la estructura de orden superior (HOS, por su sigla en inglés) empleada para respaldo de la fabricación, la comparabilidad biofarmacéutica y las evaluaciones de biosimilitud. Además, la caracterización de la HOS de las proteínas se está convirtiendo en un procedimiento "esperado" en las presentaciones regulatorias para medicamentos biofarmacéuticos y productos biosimilares.
Debido a la naturaleza compleja de las proteínas, las herramientas biofísicas son importantes para realizar una caracterización completa de un producto biofarmacéutico. Existe una variedad de herramientas biofísicas que se usan para evaluar la estabilidad de las proteínas, incluidos el (pero no limitado a) dicroísmo circular (CD, por sus siglas en inglés), la dispersión de luz dinámica (DLS, por sus siglas en inglés) y estática (SLS, por sus siglas en inglés), la cromatografía por exclusión de tamaño-dispersión de luz multiangular (SEC-MALS, por sus siglas en inglés), la espectroscopía infrarroja transformada de Fourier (FTIR, por sus siglas en inglés), la ultrafiltración de análisis (AUC, por sus siglas en inglés), cromatografía por exclusión de tamaño (SEC), fluorescencia de barrido diferencial (DSF, por sus siglas en inglés), fluorescencia intrínseca (IF, por sus siglas en inglés) y calorimetría de barrido diferencial (DSC, por sus siglas en inglés).
Si bien todos estos ensayos biofísicos desempeñan un papel importante en el desarrollo biofarmacéutico, la caracterización de la estabilidad térmica por medio de la DSC es fundamental. En un artículo publicado en 2015 sobre las técnicas biofísicas utilizadas para la caracterización de estructuras de orden superior de anticuerpos monoclonales, Gokarn et al. afirmaron: "la DSC sigue siendo una técnica inigualable para evaluar la estabilidad termodinámica de proteínas en una condición de solución amortiguadora determinada"[1].
El propósito de este documento es explicar cómo se usa la DSC para caracterizar la estabilidad térmica de los productos biofarmacéuticos con base proteica (principalmente anticuerpos) durante la selección de candidatos para medicamentos para pasar al desarrollo.
La DSC es una técnica de microcalorimetría que se emplea para caracterizar la estabilidad térmica y conformacional de proteínas, ácidos nucleicos, lípidos y otros biopolímeros [2-7]. La DSC mide la capacidad térmica como una función de temperatura. Los instrumentos de DSC empleados para la caracterización de proteínas que se describen en este documento son instrumentos de "compensación de energía", con el biopolímero en solución en una celda fija para muestras y una solución amortiguadora correspondiente dentro de una celda de referencia compatible. La señal de capacidad térmica (Cp) de la celda para muestras se compara con la de la celda de referencia. A medida que aumenta la temperatura de las celdas, las diferencias de temperatura entre la celda de referencia y la celda para muestras se miden y calibran continuamente respecto de las unidades de energía. La DSC es "un ensayo de degradación forzada", ya que la proteína se expone a una temperatura en aumento y comienza a desplegarse, mientras que la Cp de la proteína aumenta (Figura 1).
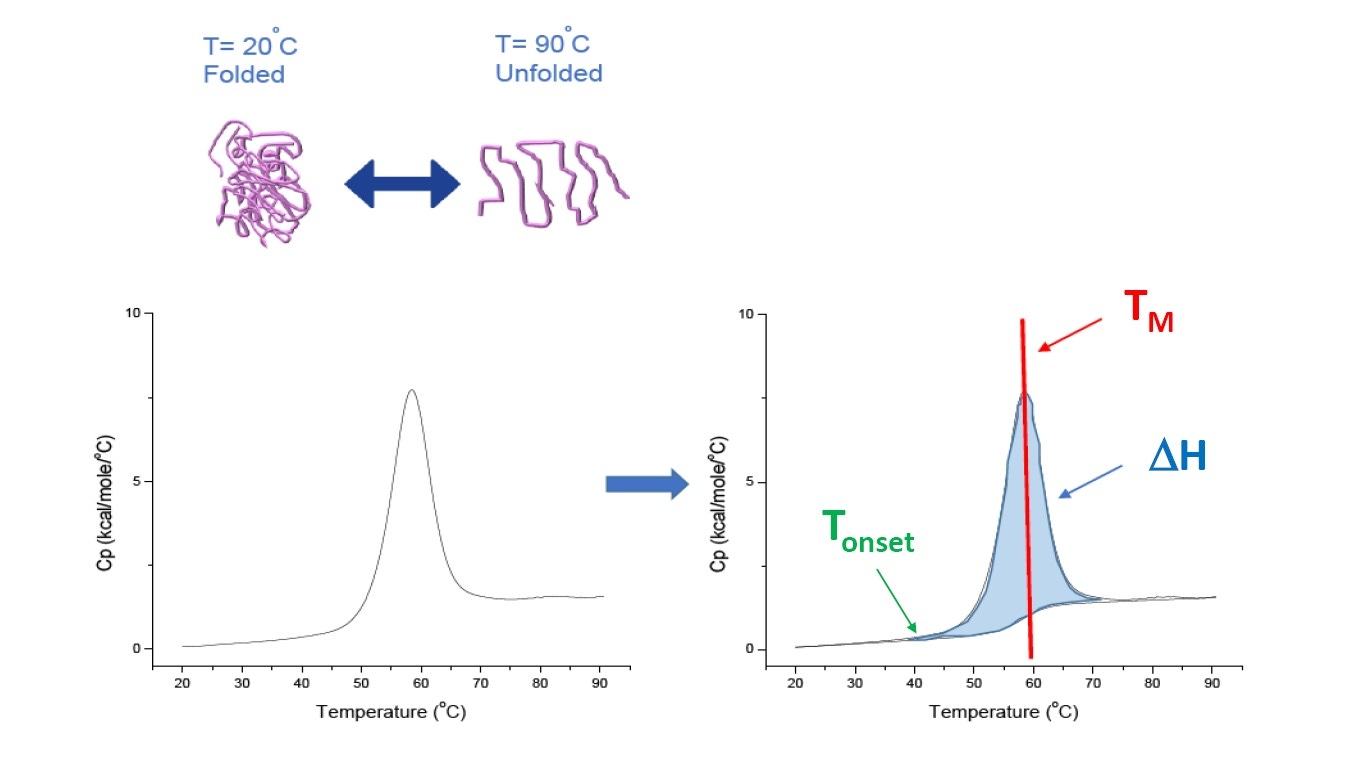
Figura 1: cómo funciona la DSC. La capacidad térmica (Cp) cambia a medida que la proteína se desnaturaliza térmicamente. El experimento de DSC comienza con una temperatura a la cual la proteína está inicialmente plegada en su conformación nativa. Con el aumento de la temperatura, en algún punto la proteína comenzará a desplegarse o desnaturalizarse (Tde inicio) y la Cp aumenta. En la temperatura a la cual el 50 % de la proteína está en su conformación nativa y el otro 50 % está desnaturalizado, la Cp alcanzará su valor máximo. Este valor es el punto medio de transición térmica o TM. Por sobre el TM, la proteína estará mayormente desnaturalizada y, al final del experimento de DSC, toda la proteína estará en su conformación desplegada. Los parámetros experimentales para la DSC incluyen la Tde inicio, el TM y la entalpía de despliegue (ΔH).
La DSC mide directamente el cambio de la capacidad térmica, sin necesidad de usar fluorescencia o cualquier otro marcador o sonda. El punto medio de transición térmica (TM), también llamado temperatura de fusión o desnaturalización, es la temperatura a la cual la proteína se encuentra con el 50 % en su conformación nativa (plegada) y el otro 50 % en su conformación desnaturalizada. El TM se observa como el "pico" en un termograma de DSC (Figura 1).
El TM se considera un buen indicador de la estabilidad térmica: mientras más alto el TM, más térmicamente estable es la proteína. Las proteínas multidominio (como los anticuerpos) habitualmente arrojan más de un pico en un termograma de DSC, de modo que se puede determinar más de un TM (consulte la Figura 2 para ver un ejemplo).
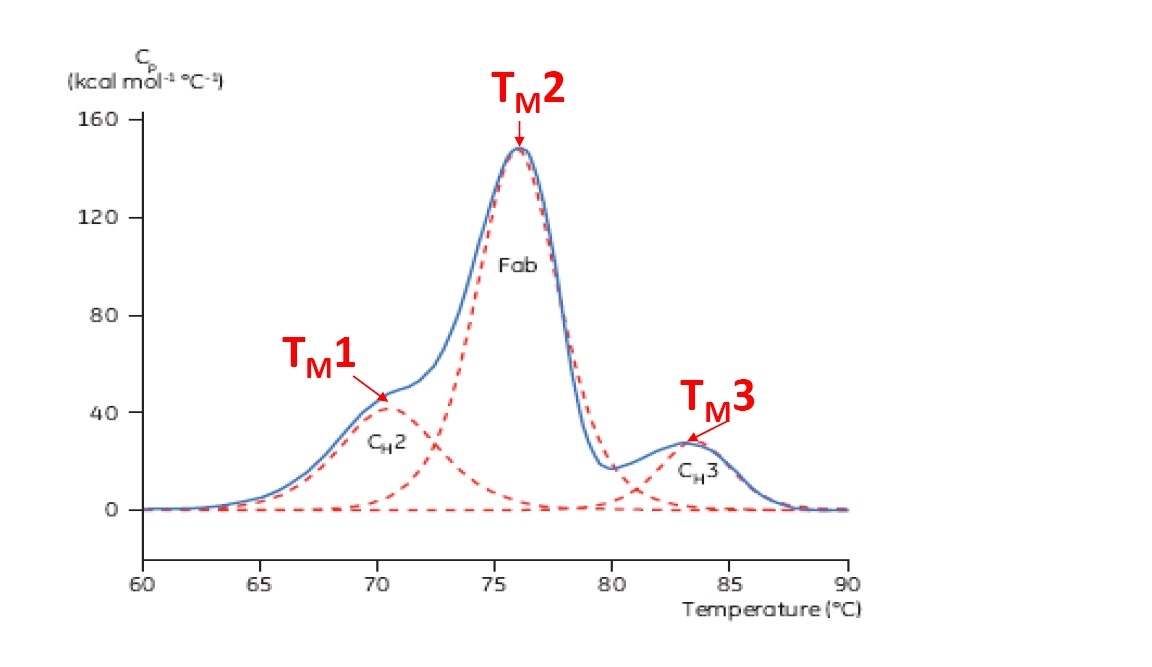
Figura 2: termograma de DSC representativo de un anticuerpo monoclonal con dominios identificados de CH2, fragmento de unión a los antígenos (Fab, por sus siglas en inglés) y CH3. Las líneas rojas discontinuas son picos deconvolucionados de cada transición de dominio con los tres TM indicados.
La DSC proporciona otros parámetros útiles que se pueden emplear para caracterizar y ordenar la estabilidad de las proteínas, incluida la entalpía de despliegue (ΔH), que se mide por el área bajo la curva. El despliegue de proteínas es un proceso endotérmico, ya que se necesita aplicar energía para romper los enlaces secundarios no covalentes que mantienen a la proteína correctamente plegada. La DSC también determina la Tde inicio (comienzo del despliegue), la ΔCp (cambio de la capacidad térmica de despliegue) y la T1/2 (ancho en la mitad de la altura del pico, muestra de la forma del termograma de despliegue). El análisis de DSC puede incluir la determinación de cualquier combinación de estos parámetros.
Malvern Instruments ofrece el sistema MicroCal VP-Capillary DSC [8,9], que corresponde a una DSC automatizada, diseñada para la detección de TM y la caracterización termodinámica de proteínas y biopolímeros en solución.
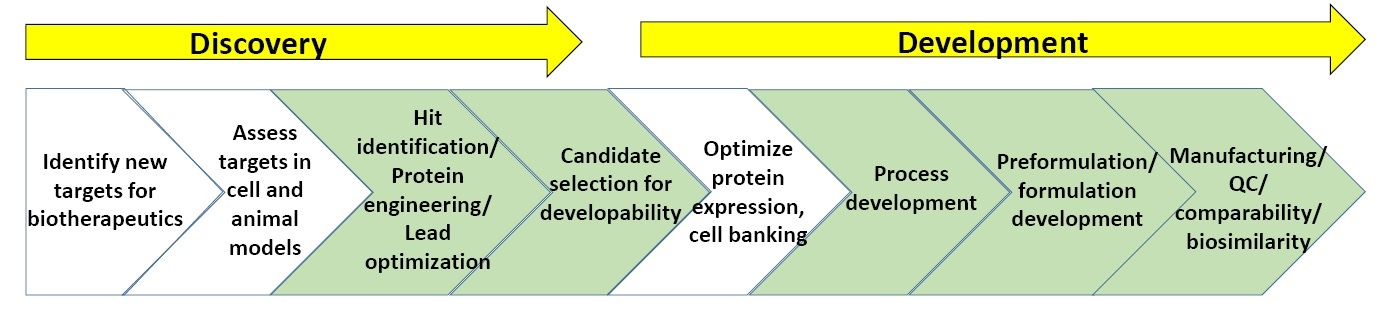
Figura 3: esquema general de los procesos en el descubrimiento y desarrollo biofarmacéutico.
La Figura 3 muestra un esquema general de los procesos de caracterización involucrados en el descubrimiento y el desarrollo biofarmacéutico. Las secciones en verde muestran dónde se usa habitualmente la caracterización biofísica, incluidos los ensayos de estabilidad. Al final de este documento hay una lista de "lecturas sugeridas" sobre el descubrimiento y el desarrollo biofarmacéutico.
La secuencia de aminoácidos o estructura primaria (1°) es el componente más básico de la cadena polipeptídica y la estructura de la proteína. Es importante conocer y caracterizar la estructura tridimensional de la proteína, también llamada estructura de orden superior (HOS). Hay tres niveles de HOS de proteína: la estructura secundaria (2°) se refiere a los patrones de plegado locales de la estructura primaria de una proteína, incluidas las hélices alfa, las láminas beta, los giros y los espirales al azar; la estructura terciara (3°) es la estructura tridimensional de una proteína que surge de su espectro de elementos de la estructura secundaria; y la estructura cuaternaria (4°), que describe las estructuras que surgen de la interacción de dos o más cadenas polipeptídicas idénticas o diferentes.
Con el fin de crear un producto biofarmacéutico apropiado, los científicos primero buscan biomoléculas que ya muestren una alta estabilidad durante la selección de candidatos y puede que sea necesario inferir un aumento de estabilidad por medio de la ingeniería de proteínas. Durante la purificación, la proteína habitualmente se aísla de condiciones donde está estable, correctamente plegada y activa, por lo que es importante usar las soluciones amortiguadoras, los aditivos, la purificación y las condiciones de almacenamiento correctos para mantener la proteína lo más estable posible durante este proceso.
Cuando las moléculas de proteína están expuestas a agentes de estrés como el calor, químicos, cambios de pH, presión, mezcla y alta concentración, que con frecuencia ocurren durante la formulación y la producción biofarmacéutica, la conformación de la proteína puede favorecer la forma desnaturalizada (desplegada). Los medicamentos subcutáneos (SC) formulados deben ser estables y no se deben ver afectados a altas concentraciones de proteínas (sobre 100 mg/mL) dentro de los cierres de su contenedor (por ejemplo, ampollas o jeringa llenada previamente), generalmente por varios años. Las proteínas en solución también son susceptibles a modificaciones como la desamidación y la oxidación, que pueden generar proteínas desnaturalizadas e inactivas.
En el caso de los productos biofarmacéuticos de base proteica, la desnaturalización y otras modificaciones pueden generar la formación de agregados que pueden provocar una eficacia reducida o una funcionalidad disminuida al medicamento. Más importante, la agregación de proteínas ha sido implicada en la generación de reacciones inmunogénicas potencialmente fatales para los pacientes. El uso de medicamentos de base proteica estables genera una producción más rentable y permite crear medicamentos más satisfactorios, eficaces y seguros.
La DSC es una medida de la estabilidad conformacional de una molécula y los cambios en la estructura terciaria y cuaternaria que se producen cuando la proteína se desnaturaliza térmicamente, así como los efectos intrínsecos y extrínsecos en la estabilidad de la proteína. La DSC se considera el análisis de mejor calidad y el más exhaustivo y cuantitativo para la caracterización de la estabilidad térmica de proteínas biofarmacéuticas y se utiliza para predecir la estabilidad a largo plazo[1,10-14]. El T M generado del uso de DSC es un parámetro que se emplea frecuentemente para ordenar la estabilidad durante la selección de candidatos (capacidad de desarrollo), las pruebas de formulación y el desarrollo del proceso. Las proteínas más estables tienen un TM más alto . La entalpía (∆H), la Tde inicio, la T1/2, y la ∆Cp obtenido con DSC se usan para ordenar la estabilidad, la validación de los datos de DSC, el análisis cuantitativo del despliegue de proteínas y la identificación de estructuras de orden superior[10-14].
Los análisis de DSC en condiciones de solución definidas son reproducibles y cuantitativos si las proteínas son las mismas o altamente similares (Figura 4). Es decir, los termogramas de DSC tendrán un patrón reproducible y los parámetros (incluidos los TM, la ∆H y la Tde inicio) estarán dentro de un rango aceptado[12-14]. Si los termogramas son diferentes y los parámetros de ajuste de DSC cambian, esto sugiere que han ocurrido eventos como el plegado incorrecto de la proteína, degradación, agregación, diferencias en la solución amortiguadora, cambios en la modificación postraduccional u otro cambio de estructura de orden superior que tienen un impacto en la estabilidad conformacional.
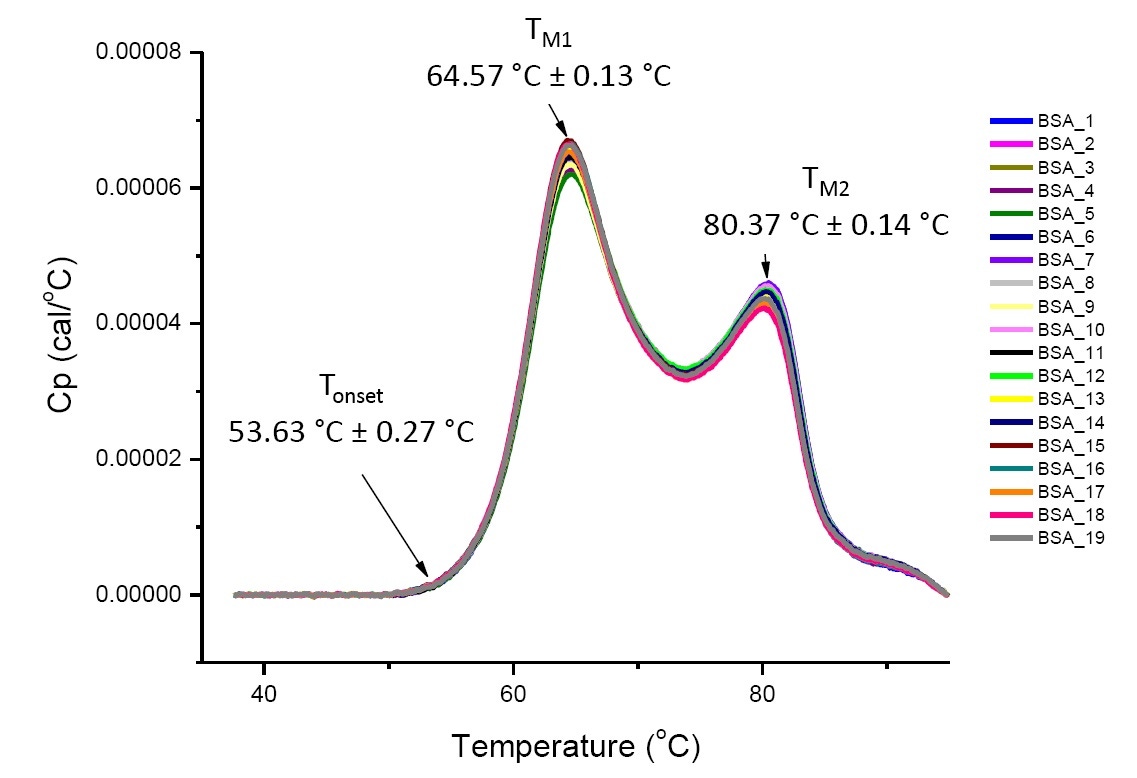
Figura 4. 19 termogramas de DSC de albúmina sérica bovina (Sigma A1933, purificado por cromatografía) en un tampón fosfato salino (PBS, por su sigla en inglés). Datos de DSC mostrados después de la normalización de la tasa de barrido, la sustracción buffer-buffer y sustracción de integración y línea de base. Se muestran las desviaciones media y estándar de la Tde inicio, el TM1 y el TM2.
Los resultados reproducibles y cuantitativos hacen de la DSC una herramienta de caracterización de HOS útil para la evaluación de productos durante la fabricación (incluidas la comparabilidad de lote con lote y sitio con sitio), la comparación de variantes de proteína y productos modificados (incluidos los cambios estructurales debido a glicosilación y oxidación) y biosimilitud. Los datos de DSC también se usan en documentos regulatorios de respaldo como una caracterización de HOS para nuevas presentaciones de medicamentos y biosimilares. En una encuesta realizada a científicos del área biofarmacéutica, la DSC fue calificada como una herramienta de análisis biofísico de HOS "muy útil" a "extremadamente útil" para la selección de candidatos, el desarrollo de formulaciones, la caracterización del producto, la comparabilidad y la biosimilitud[15].
Para las proteínas multidominio como los anticuerpos, los termogramas de DSC muestran más de una transición de despliegue (Figura 2). Con la DSC se pueden caracterizar y cuantificar los diferentes dominios y se pueden determinar los TM para dos, tres o más transiciones. Los valores de TM se pueden extraer de los picos del termograma y se pueden determinar de manera muy sencilla a partir de los datos de DSC, sin necesidad de realizar un análisis de datos complejo. Otros ensayos biofísicos que pueden determinar el TM, como el CD, la IF y la DSF, pueden detectar solo el primer TM (que ocurre a la temperatura más baja) o el TM más "dominante" para proteínas multidominio. La extracción de más de un TM de datos espectroscópicos o por fluorescencia requiere del ajuste de datos complejos y es posible que no sea reproducible.
La DSC requiere generalmente más muestra de proteína por barrido y puede presentar un menor desempeño en comparación con otros ensayos de pruebas de TM. Si la muestra es limitada, una opción sería realizar un orden inicial del TM, ya sea con DSF o IF, y luego seleccionar varias muestras para validar el TM usando DSC. Es importante validar los resultados de TM con DSC y no usar la fluorescencia o la espectrometría para el TM como único respaldo para ensayos de estabilidad. Los ensayos basados en fluorescencia pueden presentar errores que interfieren con los resultados y es posible que los resultados de TM cambien a un valor más alto (o más bajo) debido a estos errores. Algunas proteínas y condiciones de solución amortiguadora no son compatibles con la fluorescencia y es posible que no se detecten diferencias de TM mediante estos métodos. Por último, la fluorescencia y la espectrometría no determinar la entalpía de calorimetría y otros parámetros termodinámicos en comparación con la DSC.
La industria biofarmacéutica considera a la DSC un ensayo de referencia de estabilidad térmica, ya que la DSC:
En este documento, otros documentos y en notas de aplicación de Malvern y publicaciones citadas se pueden encontrar ejemplos de cómo se utilizan los datos de DSC en el descubrimiento y el desarrollo biofarmacéutico.
En la fase de investigación de la identificación de un producto biofarmacéutico de base proteica, el foco inicial está en el descubrimiento de la proteína óptima para la potencial interacción del medicamento. Una vez que se identifica el objetivo óptimo, el trabajo se enfoca en encontrar un medicamento que interactúe con este objetivo, el cual provocará un efecto biológico deseado que puede generar un resultadoclínico favorable de reducción o eliminación de un estado de enfermedad. Después de identificar uno o más candidatos para medicamentos, es importante evaluar la "capacidad de desarrollo" de estos posibles candidatos biofarmacéuticos.
La capacidad de desarrollo de un medicamento de base proteica es una evaluación de las propiedades biofísicas de las proteínas para predecir qué proteína será capaz de soportar los factores de estrés que probablemente enfrentará durante la producción del cultivo de células, la purificación, formulación, empaque, envío y el almacenamiento a largo plazo (estabilidad). Es importante entender:
Es importante realizar un análisis de la capacidad de desarrollo de un candidato para medicamento de base proteica al final del "descubrimiento" y antes de que comience el desarrollo clínico previo, dado que, una vez que se selecciona el candidato más posible y el trabajo comienza a enfocarse en avanzar en la producción, cualquier intento de cambiar el medicamento o el proceso provocará el aumento de los costos e incrementará la probabilidad de que se abandone el proyecto. Se deben tomar buenas decisiones en este punto temprano del proceso con el fin de evitar fallas, favorecer la reducción significativa de los costos para la empresa farmacéutica, así como la producción de medicamentos rentables.
En este punto en el descubrimiento tardío/el desarrollo temprano, con frecuencia hay una cantidad limitada de proteína purificada disponible para los ensayos biofísicos. También se presenta una cantidad de tiempo limitada para la evaluación del ensayo de capacidad de desarrollo. Habitualmente, en este punto se emplean métodos biofísicos como la fluorescencia, la dispersión de luz, la cromatografía por exclusión de tamaño y la DSC. Otros ensayos como la ultracentrifugación de análisis (AUC) y el intercambio de hidrógeno-deuterio con espectrometría de masa (HDX-MS, por sus siglas en inglés) también pueden entregar información útil.
La elaboración simple del perfil del medicamento de base proteica en una etapa temprana usando DSC para ordenar los valores de TM puede ser muy útil en la comparación de candidatos de medicamentos para encontrar proteínas que pueden tener una estabilidad óptima, para predecir la estabilidad a largo plazo y para evitar que esas moléculas que tienen menos estructuras térmicamente estables. Las proteínas con los valores de TM más bajos son considerados los menos apropiados para progresar como candidatos para medicamentos, ya que tienen una mayor inestabilidad como una función de temperatura.
Doyle et al.[16] publicaron un estudio de caso utilizando métodos biofísicos para caracterizar candidatos de adnectina biespecífica durante el descubrimiento, incluida la selección de candidatos. Los ensayos biofísicos incluyeron: SEC, AUC, SEC-MALS, cristalografía de rayos X, resonancia de plasmones superficiales (SPR; por sus siglas en inglés), DSF y DSC. La comparación de dos candidatos de adnectina mediante DSC arrojó que el candidato B mostró valores de TM y Tde inicio más altos (ambos indican una mayor estabilidad térmica) en comparación con el candidato A. El candidato B también mostró evidencia de reversibilidad de plegado con DSC. Esto significa que la muestra de proteína se enfrió y se volvió a calentar en la DSC, y el 2dociclo de calentamiento arrojó el mismo perfil de DSC. La reversibilidad también indica que existen niveles de expresión mejorados y una tendencia reducida a la formación de agregados.
La clase de proteína de mayor tamaño de productos biofarmacéuticos en el mercado o en pruebas clínicas son anticuerpos monoclonales. Los anticuerpos monoclonales habitualmente arrojan un termograma complejo multidominio en la DSC (consulte la Figura 2 para ver un ejemplo). El pico de dominio más grande y prominente es el Fab (fragmento de unión a los antígenos) del anticuerpo. Los dominios CH2 o CH3 también se observan de manera habitual. Las posiciones relativas y los TM de los diferentes dominios dependen de los anticuerpos monoclonales específicos y pueden variar según la subclase y el diseño, como se demuestra en los ejemplos que se entregan más abajo.
En 2007, Garber y Demarest publicaron un artículo que describe el uso de DSC para caracterizar 17 anticuerpos terapéuticos de longitud completa[17,18]. Con el uso de MicroCal VP-Capillary DSC, los Fab de estos anticuerpos mostraron transiciones de despliegue térmico con puntos medios (TM) que iban de 57 °C a 82 °C. Los autores demostraron que la IgG1 fue la subclase humana más estable, seguida de IgG2 y IgG4, y para los ejemplos utilizados, mostraron que las variaciones de estabilidad entre anticuerpos de subclases similares se derivaron de dominios variables. Las características únicas de cada gen V generaron una amplia gama de estabilidades de Fab.
Algunos Fab de anticuerpos pueden ser excepcionalmente estables, mientras que otros pueden ser relativamente inestables. La considerablemente baja estabilidad de Fab que se dedujo del TM, como se observó en varios constructos incluidos en la referencia 17, se relacionó con una expresión baja o reducida del anticuerpo y con un aumento de problemas de agregación en comparación con los anticuerpos con mayor TM de Fab. Como resultado, es más difícil purificar proteínas con un TM más bajo, lo que genera un desempeño reducido durante el desarrollo del proceso, así como una mayor probabilidad de agregación durante el almacenamiento. Los datos obtenidos de este estudio[17] entregaron evidencia de que los datos de estabilidad de DSC son una herramienta útil para la selección de candidatos para medicamentos biofarmacéuticos.
Ionescu et al.[19] generaron perfiles de DSC (con MicroCal VP-Capillary DSC) de tres anticuerpos monoclonales IgG1 humanizados y sus Fab y fragmentos cristalizables (Fc) (luego de la digestión enzimática) en un pH neutro. Con algunas excepciones, los termogramas mostraron que la transición con entalpía experimental más grande contenía la contribución de fragmentos Fab (similar a los resultados en la referencia 17). Además, se encontró que los TM aparentes variaron significativamente, incluso para fragmentos Fab que originaron la misma célula germinal humana. Ionescu at al. propusieron el uso de la entalpía de despliegue como el parámetro clave para reconocer los eventos de despliegue en el perfil de derretimiento en un anticuerpo IgG1 intacto. Algunos termogramas de DSC mostraron dos transiciones: la primera transición, que representaba el despliegue del fragmento Fab y el dominio CH2; y la segunda transición, que representaba el despliegue del dominio CH3. En otros anticuerpos, la primera transición de la DSC representa el despliegue del dominio CH2 y la segunda transición representa el despliegue del fragmento Fab y del dominio CH3. Hubo otros casos donde el perfil de DSC presentó tres transiciones con el despliegue de Fab que ocurrieron a distintas temperaturas en comparación con el derretimiento de los dominios CH2 y CH3. La baja estabilidad o heterogeneidad del fragmento Fab puede suponer un problema para el almacenamiento a largo plazo o la consistencia de la producción. Los autores propusieron que el conocimiento acabado de las características de un perfil de DSC es importante para la selección de candidatos en etapas tempranas del desarrollo de anticuerpos monoclonales terapéuticos.
El trabajo descrito por los dos artículos anteriores, así como otros estudios publicados durante el mismo período, formaron los fundamentos para los estudios de caso posteriores y el uso de DSC y otros estudios de caracterización de estabilidad como parte de la selección de candidatos biofarmacéuticos.
Para delinear con más detalle la estabilidad térmica de candidatos para medicamentos, las proteínas se analizan frecuentemente en más de una condición de pH para ver si surgen diferencias. Estas diferencias de pH pueden representar condiciones que se observan en formulaciones o soluciones amortiguadoras de procesos empleados durante la fabricación de los medicamentos.
Jiang et al.[20] publicaron un estudio de caso donde se usó la caracterización de HOS seleccionada para evaluar la capacidad de desarrollo (capacidad de fabricación, así como de mantención de la calidad general del producto) de dos potenciales medicamentos de anticuerpos monoclonales. Los anticuerpos X e Y fueron los mAb IgG2 contra el mismo objetivo, lo cual mostró actividades biológicas similares. Usando una combinación de técnicas biofísicas (incluidas la DSC, CD UV cercano, FTIR, DLS, espectroscopía de fluorescencia y SEC), observaron:
Se seleccionó la condición ácida (pH 3) para replicar las soluciones amortiguadoras utilizadas durante la purificación de anticuerpos y la inactivación viral. Durante la purificación, se usaron condiciones de almacenamiento simulado con una solución amortiguadora de pH 5 y una solución amortiguadora de pH 7.
Los barridos con MicroCal VP-Capillary DSC de los dos candidatos mAb en una solución amortiguadora con pH 7 (PBS) mostraron dos transiciones: los dominios CH2/Fab representaron la 1era transición, mientras que el despliegue del dominio CH3 estuvo representado por la 2da transición[20]. El mAb Y tuvo una estabilidad térmica levemente superior en el pH 7, sobre la base de los TM obtenidos de la DSC, así como una mayor temperatura de inicio (Tde inicio). En la solución amortiguadora de pH 3 (C3N), hubo solo una transición térmica de DSC para cada candidato, lo que sugiere que las muestras ya estaban parcialmente desplegadas a la temperatura inicial debido al pH bajo. Las temperaturas de transición térmica y las entalpías del despliegue de ambos candidatos en el pH 3 fueron notoriamente inferiores a aquellos en PBS, como se esperaba. Sin embargo, el mAb X (TM de 63,2 °C) mostró una mejor estabilidad térmica que el mAb Y (TM de 48,2 °C) en C3N, a pesar de que el mAb Y fue térmicamente más estable en pH neutro. Aunque este resultado es inusual, es coherente con los datos de CD UV cercano, que mostraron que el mAb X retuvo más estructuras terciarias en el pH 3. Los cambios en la estabilidad térmica de los dos candidatos inducidos por el tratamiento con pH 3 resultaron ser completamente reversibles luego de la diálisis en PBS.
En general, los resultados de la DSC indicaron que ambas proteínas eran muy estables en un pH neutro y que la estabilidad térmica del mAb Y fue mayor que la del mAb X en PBS, pero menor que la del mAb X en C3N[20]. Los datos de CD y DSC mostraron que el mAb Y sufrió más cambios estructurales en el pH 3 que el mAb X. Los cambios de estabilidad térmica inducidos por un pH 3 eran completamente reversibles para ambos mAb, aunque los cambios de estructura terciaria parecieron ser, al menos, parcialmente irreversibles para el mAb y. En comparación con el mAb X, el mAb Y mostró una mejor estabilidad térmica en un pH neutro y mejoró la estabilidad de almacenamiento a 37 °C en la solución amortiguadora de su formulación con pH 5. En conjunto, los resultados sugieren que hay más fuerzas que impulsan la estabilidad del proceso (consistencia con cambios de pH) y la estabilidad de almacenamiento (impacto de una temperatura elevada).
Dado que el mAb Y plegado tuvo una estabilidad térmica mayor en PBS con un pH 7 y una mejor estabilidad con un pH 5 durante el almacenamiento a 37 °C, se recomendó que mAb Y debía avanzar para continuar con el desarrollo[20]. Además, debido a la irreversibilidad de la disrupción de la estructura terciara y la autoasociación en PBS que se observó luego de una incubación con pH bajo, la recomendación de proceder con mAb Y fue acompañada por una recomendación para desarrollar el proceso de purificación para minimizar la exposición de mAb Y con un pH 3. Este estudio de caso demostró cómo los datos de estabilidad como parte de la selección de candidatos y el desarrollo del proceso ayudan a minimizar la cantidad de agregados irreversibles y el variante conformacional producido por el proceso y, básicamente, aumentarán el rendimiento del proceso[20].
Varios otros estudios de caso que usaron la estabilidad térmica con DSC para evaluar y predecir la capacidad de desarrollo se resumen en Satish et al.[21]
Tavakoli-Kenshe et al.[22] quisieron observar qué otros factores predictivos, además de los estudios de estabilidad, se pueden emplear para predecir la capacidad de desarrollo. Se observaron cinco variantes de los anticuerpos IgG1 y IgG4 y utilizaron un dispositivo de corte de disco giratorio, que produjo condiciones de corte definidas en una interface sólida-líquida conocida para medir la estabilidad en este ambiente. Los anticuerpos se ordenaron según la estabilidad en función del resultado del dispositivo de corte y en comparación con los datos de estabilidad térmica acelerada y la temperatura de fusión del dominio CH2 (TM1) obtenidos con MicroCal VP-Capillary DSC para investigar la complementariedad de la técnica. Los resultados sugirieron que las técnicas son ortogonales con métodos térmicos basados en la interacción intramolecular, y la estabilidad del dispositivo transversal en función del despliegue localizado, lo que revela menos regiones estables que impulsan la agregación. El modelo molecular muestra los efectos de las modificaciones en las estructuras de los anticuerpos e indica que hay un posible papel de la conformación de Fc y el acoplamiento de Fab-Fc en la determinación de la estabilidad de proteínas suspendidas. Los datos introducen una prueba de adaptación como un potencial indicador de estabilidad ortogonal, que complementa los métodos térmicos tradicionales como la DSC, lo que permite que la selección de candidatos de proteínas tenga una estabilidad mejorada para el proceso de purificación.
Habitualmente en el descubrimiento biofarmacéutico, las moléculas "prototipo" o moléculas "originales" de medicamentos de base proteica se modifican o diseñan para mejorar sus características biofísicas y hacer más desarrollable la proteína. Luego del diseño y antes de la selección de candidatos, es importante evaluar la estabilidad de las diferentes proteínas diseñadas para ver si el diseño tuvo algún efecto perjudicial. Un ejemplo de esto se presentó una nota de aplicación de Malvern[23], donde se usó MicroCal VP-Capillary DSC como una herramienta de prueba para el anticuerpo original y dos anticuerpos diseñados, para ayudar a predecir la estabilidad térmica y la capacidad de desarrollo de los constructos diseñados. También se estudió la formación de agregación (a través de SEC) luego del almacenamiento acelerado. El anticuerpo diseñado con el TM que más disminuyó se correlacionó con el constructo del anticuerpo con la mayor cantidad de agregación luego de la prueba de estabilidad acelerada, lo que permitió obtener una orientación de la selección del candidato.
Demarest et al. diseñó un Fab inestable y utilizó MicroCal VP-Capillary DSC para observar la estabilidad de la proteína como una demostración[18, 24]. En un esfuerzo por desarrollar estrategias de estabilización de anticuerpos y fragmentos de anticuerpos, seleccionaron un Fab de mal comportamiento que reconoció la toxina tetanus (αTT) originalmente aislada de una fuente humana, y se realizó una mutagénesis para diseñar la estabilidad en el Fab. Los autores seleccionaron 45 posiciones de residuo para aleatorización y se seleccionaron y cultivaron cerca de 4500 colonias individuales en medios de expresión. Los sobrenadantes que contenía el Fab expresado se expusieron a calor con tres temperaturas elevadas (70 °C, 72 °C y 74 °C). Las variantes que demostraron una termoestabilidad mejorada fueron sometidos nuevamente a la prueba de termotolerancia para confirmar sus propiedades (consulte la referencia 24 para conocer una lista completa de mutantes estabilizadores). Aproximadamente el 1% de las variantes en la biblioteca mostraron una termoestabilidad mejorada. Catorce de las moléculas prototipo se ubicaron en el dominio VH y las cuatro restantes en el dominio VL. Este resultado sugiere que la estabilidad del Fab nativo se vio limitado por la estabilidad marginal de VH. Sorprendentemente, ningún mutante dentro de las bibliotecas de dominio constante de aproximadamente 2000 elementos mostró una estabilización del Fab en su totalidad. Los autores especularon que las mutaciones estabilizadoras dentro de los dominios constantes realmente ocurren, pero que la estabilidad limitada del dominio VH controla la temperatura a la cual se despliega el Fab y limita la capacidad de observar estos eventos.
Se descubrieron cuatro mutantes estabilizadores dentro del dominio VL de αTT[24]. La mutación del residuo consenso VK4 W50 a Ala (el residuo de consenso para VK1) o His fue altamente estabilizadora. La histidina no se encuentra habitualmente en la posición 50 en los dominios variables kappa humanos, pero se encuentra con frecuencia en los dominios variables lambda humanos. Este residuo está cerca de la interface del dominio VH/VL. Los autores tenían la hipótesis de que su contribución a la estabilidad de Fab podría estar relacionada con el reforzamiento del dominio VH, ya que el dominio VH en particular parece limitar la estabilidad del Fab de la αTT. Sin embargo, los estudios con el dominio VL aislado mostraron lo contrario.
Se generaron doce constructos con un contenido de entre tres a once mutaciones estabilizadoras que se identificaron en la prueba inicial[24]. Se derivaron racionalmente varias combinaciones para determinar la supuesta contribución de cada mutante a la estabilización del Fab. La introducción de mutaciones estabilizadoras múltiples al Fab de la αTT aumentó la expresión del Fab en un experimento de transformación/expresión paralelo, lo que sugirió que los constructos más estables generaron un aumento de la expresión en las células. Los mejores constructos mostraron de manera consistente más de tres aumentos de despliegue en el desempeño expresado en comparación con el tipo natural. Se evaluó la estabilidad de cada Fab por medio de DSC y dicroísmo circular (CD). Los valores de TM medidos se usaron para ordenar la supuesta estabilidad del Fab.
Una consideración importante fue el potencial efecto que podrían tener estas mutaciones en el enlace del antígeno funcional del Fab de la αTT. Las mutaciones termoestabilizadoras se derivaron de la prueba original usando ELISA cuantitativo, lo que detectó el dominio CL y la etiqueta de histidina en el extremo C de CH1. Los autores observaron que las mutaciones termoestabilizadoras mejoraron la aparente afinidad del Fab de la αTT en un ensayo ELISA funcional, en comparación con el tipo natural[24]. La capacidad funcional de cada variante de Fab pareció estar directamente relacionada con su estabilidad, como lo describe el TM obtenido de la DSC. La capacidad de correlacionar la actividad funcional y la expresión de la proteína con el TM obtenido de la DSC sugirió que elegir el constructo más estable mejoraría la capacidad de desarrollo.
Seeliger et al.[25] incorporaron las estrategias de diseño computacional para modificar sistemáticamente un anticuerpo que exhibió una tendencia a generar agregados in vitro. La serie resultante de anticuerpos estrechamente relacionados diseñados a partir de un mAb1 de tipo natural (WT, por sus siglas en inglés) mostró una estabilidad mejorada, según lo evaluado con los métodos biofísicos (incluido MicroCal VP-Capillary DSC), así como con experimentos de estabilidad a largo plazo. Como Demarest et al. constataron en el ejemplo anterior con el Fab de la αTT[24], los niveles de expresión de la proteína mutante también mejoraron en comparación con el candidato de tipo natural. En conjunto, los datos experimentales y computacionales empleados en este estudio demuestran cómo se pueden emplear los métodos computacionales para orientar la optimización de anticuerpos para una estabilidad mejorada.
La DSC de la proteína tipo natural original mostró un TM de 68 °C en la primera transición del termograma (identificado como el dominio Fab). En termogramas de DSC para mutaciones de cadena pesada o cadena ligera, el TM de la primera transición térmica (Fab) de DSC aumentó a 68,9 °C - 72,8 °C. En el mutante con cadenas pesadas y ligeras modificadas, la primera endotermia se observó a 70,5 °C, con un su mayor pico (Fab) a 83,5 °C. El TM para el despliegue del Fab del anticuerpo aumenta aproximadamente 16 K después de la extracción de los "elementos responsables" que se predijeron en la cadena pesada y la cadena ligera. Cuando se monitoreó el despliegue a través del cambio en la fluorescencia de los triptófanos, se observó la misma tendencia[25].
La simulación computarizada predijo que la mayoría de las mutaciones introducidas aumentan la estabilidad termodinámica. Estos resultados concordaron con los obtenidos en las mediciones de DSC y la dispersión de luz de ángulo derecho (RALS, por sus siglas en inglés). Ambos experimentos demostraron un aumento continuo en la estabilidad con un número creciente de mutaciones. En ambos experimentos, los autores también descubrieron que el diseño de la cadena ligera tuvo en efecto notorio en la estabilidad, lo que concuerda con los cambios calculados de la termoestabilidad para cada mutación[25].
Todas las variantes diseñadas de mAb1 se expresaron en dosis sobre 50 mg/L, mientras que con el tipo natural, las dosis de mAb1 estuvieron bajo 1 mg/L. No se puso predecir el aumento significativo de los niveles de expresión para las variantes con solo una cadena diseñada a partir de los resultados de DSC, lo que mostró un aumento pequeño de la estabilidad para estas variantes. Sin embargo, los cálculos sugieren que la estabilidad de los dominios de inmunoglobulina individuales mejora con las mutaciones introducidas. En experimentos con calor, es evidente que la estabilidad de cada cadena individual es el factor limitante, lo que indica que los efectos observados fueron más bien cinéticos que termodinámicos de equilibrio. Tan pronto se despliega un dominio, el efecto estabilizador de la formación del complejo desaparece, y, como consecuencia, la segunda cadena comienza a desplegarse también. Para la producción y plegado de proteínas in vivo, la presencia de un dominio plegado estable para servir como un punto de nucleación para una cadena menos estable puede ser suficiente para aumentar significativamente la cantidad de anticuerpos exitosamente plegados[25].
Desde la perspectiva de una empresa que desarrolla un medicamento biofarmacéutico, ¿el aumento escalonado de la estabilidad de la proteína (a partir de DSC y otros ensayos biofísicos) se traduce en un crecimiento escalonado a largo plazo y una mejor vida útil? Los autores realizaron un estudio acelerado de estabilidad a 40 °C con las variantes diseñadas y observaron mejoras significativas en la estabilidad a largo plazo, según lo concluido del contenido de monómeros a partir de SEC. La variante completamente diseñada también fue más estable en todo el período de 4 meses de la prueba[23]. La estabilidad termodinámica de los dominios variables es sin duda un factor importante para la estabilidad a largo plazo. Pero es también evidente, a partir de los datos de los autores y otros datos publicados, que la estabilidad termodinámica y la vida útil no se pueden correlacionar fácilmente en cada caso y puede que sea necesario hacer ajustes para mejorar la estabilidad en la fase de formulación[25,26,27,28].
Las publicaciones recientes describen el diseño racional y el uso de DSC y otras herramientas biofísicas para caracterizar la estabilidad de las proteínas, que incluyen:
Se evaluó el material de referencia[35] del anticuerpo monoclonal del Instituto Nacional de Estándares y Tecnología (NISTmAb, por sus siglas en inglés) para analizar su capacidad de desarrollo con herramientas biofísicas de HOS habitualmente empleadas para candidatos para medicamentos de mAb. El NISTmAb no está diseñado para ser una molécula terapéutica y la evaluación de la capacidad de desarrollo del NISTmAb se abordó como si se tratara de un mAb IgG1 representativo específico de su clase[36]. El NISTmAb se evaluó en diferentes condiciones de solución amortiguadora en términos de estabilidad e integridad usando métodos de electroforesis, SEC-MALS, DLS y DSC. La estabilidad térmica de la muestra de NISTmAb en una solución amortiguadora de PBS se determinó por medio de MicroCal VP-Capillary DSC. La muestra de NIST arroja tres transiciones térmicas principales asignadas como el dominio CH2 a 71,2 °C, el dominio CH3 a 84,1 °C y el dominio Fab a 88,9 °C.
En comparación con los perfiles de DSC de moléculas IgG1 de la referencia 17, el Fab de NIST es más estable (88,9 °C). Por esta razón, se concluyó que la molécula tiene un perfil de HOS favorable[36]. Sobre la base del paquete completo de datos de análisis biofísicos, el NISTmAb tiene un perfil de capacidad de desarrollo favorable y los cambios observados en las moléculas luego de la exposición a condiciones de estrés no representan riesgos que no se puedan mitigar con la formulación o la fabricación correcta en etapas más avanzadas del proceso de desarrollo.
Los resultados presentados en este documento demuestran con claridad la importancia y la efectividad de incorporar la DSC como un ensayo de estabilidad biofísico durante la selección de candidatos biofarmacéuticos. Con los resultados de DSC, junto con los de otros ensayos de estabilidad, las empresas biofarmacéuticas pueden tomar decisiones informadas sobre los candidatos para medicamentos más estables y desarrollables, lo que incluye a aquellos con la mejor probabilidad para la producción y purificación y aquellos con menos probabilidad de exhibir problemas de estabilidad a largo plazo y agregación para la formulación final y el medicamento. Esto se traduce en una producción de medicamentos más rentable y una mayor probabilidad de que la formulación final del medicamento se mantenga activa, estable, segura y en una conformación correctamente plegada.
Analytical Techniques for Biopharmaceutical Development, R. Rodriguez-Diaz, T. Wehr, S. Tuck (ed.), Taylor & Francis, Nueva York EE. UU. (2005).
Biophysical Characterization of Proteins in Developing Biopharmaceuticals, D.J. Houde, S.A. Berkowitz (ed.), Elsevier, Amsterdam, Países Bajos (2015).
Biophysical Methods for Biotherapeutics: Discovery and Development Applications, T.K. Das (ed.) John Wiley & Sons, Hoboken Nueva Jersey EE. UU (2014).
Biophysics for Therapeutic Protein Development, L.O. Nahri (ed.), Springer Nueva York, EE. UU (2013).
State-of-the-Art and Emerging Technologies for Therapeutic Monoclonal Antibody Characterization Volumen 1. Monoclonal Antibody Therapeutics: Structure, Function, and Regulatory Space, J.E. Schiel, D.D. Davis, O.V. Borisov (ed.), ACS Symposium Series Volumen 1176 (2014). doi: 10.1021/bk-2014-1176.
State-of-the-Art and Emerging Technologies for Therapeutic Monoclonal Antibody Characterization Volumen 2. Biopharmaceutical Characterization: The NISTmAb Case Study, J.E. Schiel, D.D. Davis, O.V. Borisov (ed.), ACS Symposium Series Volumen 1201 (2015). doi: 10.1021/bk-2015-1201.
State-of-the-Art and Emerging Technologies for Therapeutic Monoclonal Antibody Characterization Volumen 3. Defining the Next Generation of Analytical and Biophysical Techniques, J.E. Schiel, D.D. Davis, O.V. Borisov (ed.), ACS Symposium Series Volumen 1202 (2015) DOI: 10.1021/bk-2015-1202.