La stabilité d’une protéine biopharmaceutique joue un rôle déterminant dans le succès ou l'échec du développement d’un médicament viable. La stabilité des protéines est importante lors de l’étape de production, de fabrication, de formulation, de conservation à long terme, d’administration du médicament au patient et pour qu’elles soient efficaces. Les protéines extrêmement stables rencontreront vraisemblablement moins de problèmes pendant le processus de fabrication, ce sont des protéines produites de manière peu onéreuse et qui ont davantage tendance à rester fonctionnelles pendant l’étape de formulation et de conservation sans subir d’altération ou d'agrégation. La définition de la qualité par la conception (ou « QbD, Quality by Design ») est une approche du développement de produits biopharmaceutiques dont la caractérisation de la stabilité constitue la première évaluation du potentiel de développement et des propriétés pharmaceutiques d’une molécule candidate. C’est un concept également utilisé lors de la phase de développement et de fabrication de procédés. Les données relatives à la stabilité sont prises en compte pour déterminer la structure d’ordre supérieur et la cartographie peptidique utilisées à des fins de fabrication, de comparabilité biopharmaceutique et de biosimilarité. Le fait de déterminer la structure d’ordre supérieur d’une protéine est devenu une pratique courante lorsqu’il s’agit de soumettre une demande d’approbation réglementaire de médicaments biopharmaceutiques et de biosimilaires.
Compte tenu de la nature complexe des protéines, les instruments capables de déterminer les propriétés biophysiques ont un rôle important dans l’analyse d’un produit biopharmaceutique. Il existe divers instruments biophysiques généralement utilisés pour évaluer la stabilité d’une protéine, notamment (mais non exclusivement) le dichroïsme circulaire (DC), la diffusion dynamique et statique de la lumière (DLS et SLS), la chromatographie d’exclusion stérique (filtration sur gel) couplée avec la mesure de la diffusion de la lumière multi-angle (SEC-MALS), la spectroscopie infrarouge à transformée de Fourier (FTIR), l'ultrafiltration à flux tangentiel, la chromatographie d’exclusion stérique (SEC), la fluorimétrie différentielle à balayage (DSF), la fluorescence intrinsèque (IF) et la calorimétrie différentielle à balayage (DSC).
Même si toutes ces techniques biophysiques jouent un rôle important dans le développement des produits biopharmaceutiques, il est indispensable d'utiliser la DSC pour déterminer la stabilité thermique. Dans un article datant de 2015 portant sur les techniques biophysiques utilisées pour déterminer la structure d’ordre supérieur d’anticorps monoclonaux, Gokarn et al. ont déclaré :« La DSC reste une technique incomparable pour évaluer la stabilité thermodynamique des protéines dans une solution tampon donnée »[1].
Cet article a pour but d’expliquer la manière dont la DSC est utilisée pour caractériser la stabilité thermique de produits biopharmaceutiques de type protéique
La DSC est une technique de microcalorimétrie utilisée pour déterminer la stabilité thermique et conformationnelle des protéines, des acides nucléiques, des lipides et d’autres biopolymères. La DSC mesure la capacité calorifique en fonction de la température. Les instruments de DSC utilisés pour caractériser une protéine et décrits dans cet article sont des instruments de « compensation de puissance » avec une cellule d’échantillonnage contenant le polymère en solution et une cellule d'étalonnage correspondante contenant le même tampon. La capacité calorifique (Cp) de la cellule d'échantillonnage est comparée à celle d’étalonnage. Au fur et à mesure que la température augmente, les différences de température relevées entre les cellules d’étalonnage et d'échantillonnage sont systématiquement mesurées et calibrées selon des unités de puissance. La DSC est un « essai de dégradation accéléré » puisque lorsque la protéine est exposée à des températures croissantes, elle se dénature et la capacité calorifique Cp de la protéine augmente (Figure 1).
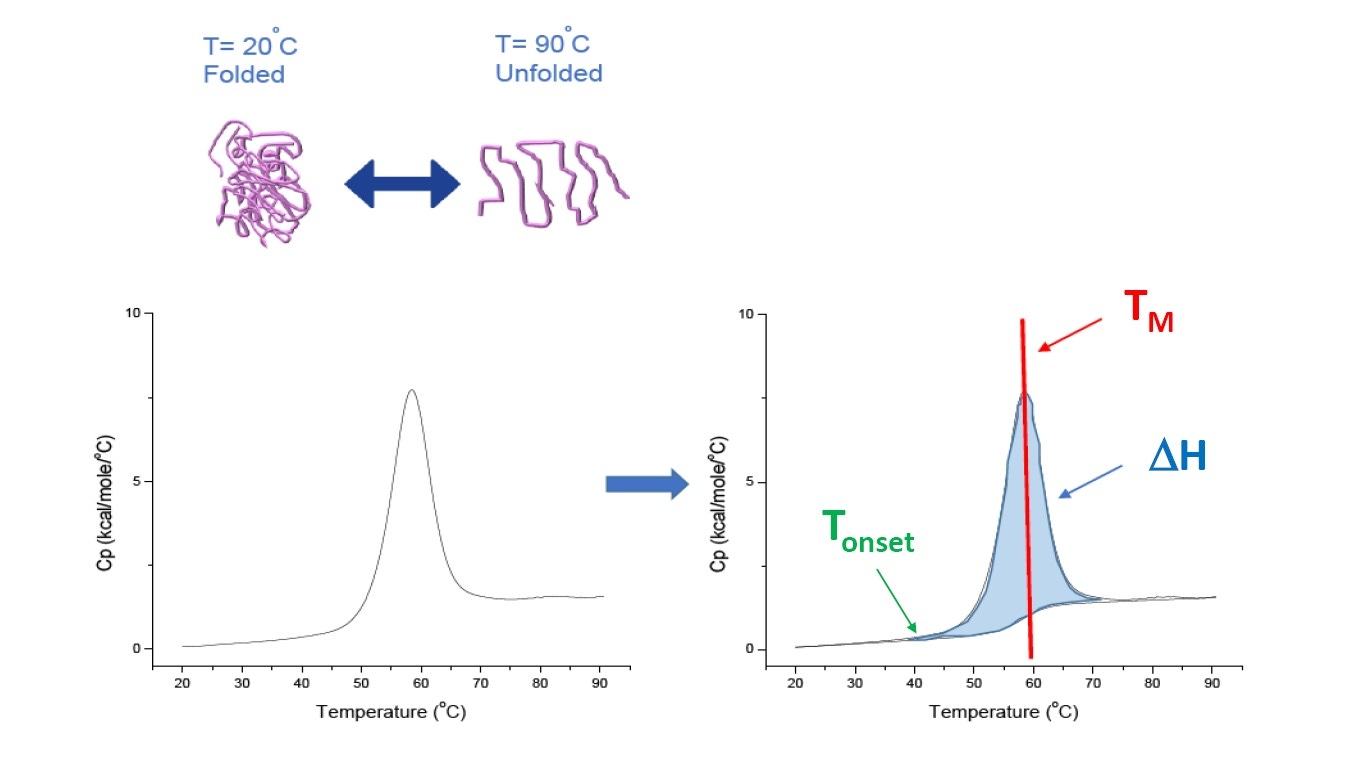
Figure 1: Comprendre le fonctionnement de la DSC: La capacité calorifique (Cp) change lorsque la protéine est dénaturée sous l’effet de la chaleur. Le test DSC débute à une température où la protéine est essentiellement à l’état natif (protéine repliée). Lorsque la température s’élève la protéine commence à se dénaturer, elle se dénature à certain endroit (Tonset) et la capacité calorifique Cp augmente. La capacité calorifique Cp atteindra sa valeur maximale à une température indiquant un équilibre entre état dénaturé et état natif de la protéine, défini par la point médian de transition thermique ou TM. Pour des valeurs supérieures à ce point TM, la protéine sera principalement dénaturée et c’est au terme du test DSC que l’on observera une dénaturation complète de l’ensemble de la protéine. Parmi les paramètres expérimentaux s’appliquant à la DSC, on trouve la Tonset, la TM et l’enthalpie de dénaturation (ΔH).
La DSC mesure directement la variation de la capacité calorifique sans avoir recours à des agents fluorescents ou à tout autre sonde ou marqueur. Le point médian de transition thermique (TM), également appelé température de fusion ou de dénaturation, correspond à la température où la protéine est à son état d’équilibre conformationnel (équilibre entre état natif et dénaturé). Le pic observé sur un thermogramme DSC correspond à la TM.
La TM est un bon indicateur de stabilité thermique. Plus elle est élevée, plus la protéine est stable d’un point de vue thermique. En général, sur un thermogramme DSC plusieurs pic apparaissent avec des protéines multidomaines telles que les anticorps. Par conséquent, il est possible d’identifier différentes TM (voir Figure 2).
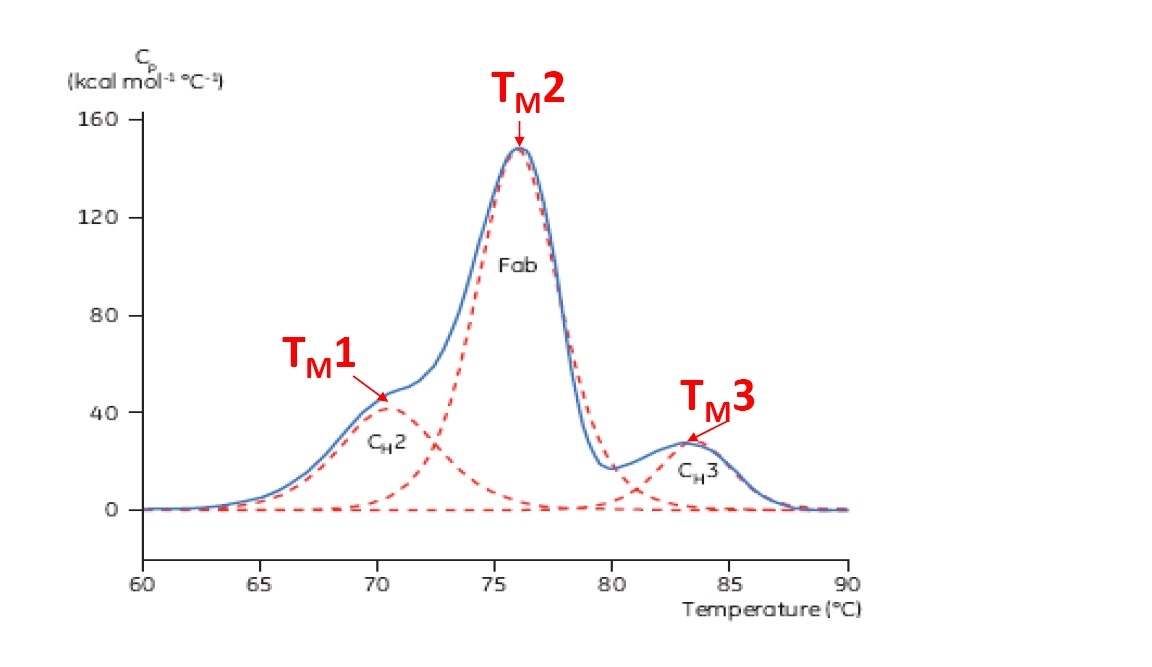
Figure 2: Thermogramme DSC représentatif d’un anticorps monoclonal constitué d’un fragment CH2, CH3 et Fab. Les lignes rouges en pointillées correspondent aux déconvolutions de pics pour chaque transition de fragments. On observe ainsi les trois points TM.
La DSC fournit d’autres paramètres importants pouvant être utilisés pour caractériser et définir la stabilité protéique, notamment l’enthalpie de dénaturation (ΔH) qui correspond à la surface sous la courbe. La dénaturation de la protéine est un processus endothermique puisque la production d’énergie est nécessaire pour rompre les liaisons secondaires non-covalentes et maintenir la protéine repliée. La DSC détermine également la Tonset (début de la dénaturation), le point ∆Cp (variation de la capacité calorifique associée à la dénaturation) et le point T1/2 (largeur à mi-hauteur du pic, révélateur de la forme du thermogramme représentatif de la dénaturation). L’analyse par DSC permet de définir plusieurs ensembles de ces paramètres.
Malvern Instruments offre le système DSC MicroCal VP-Capillary qui est un calorimètre différentiel à balayage conçu pour analyser la TM et permettre la caractérisation thermodynamique de protéines et de biopolymères en solution.
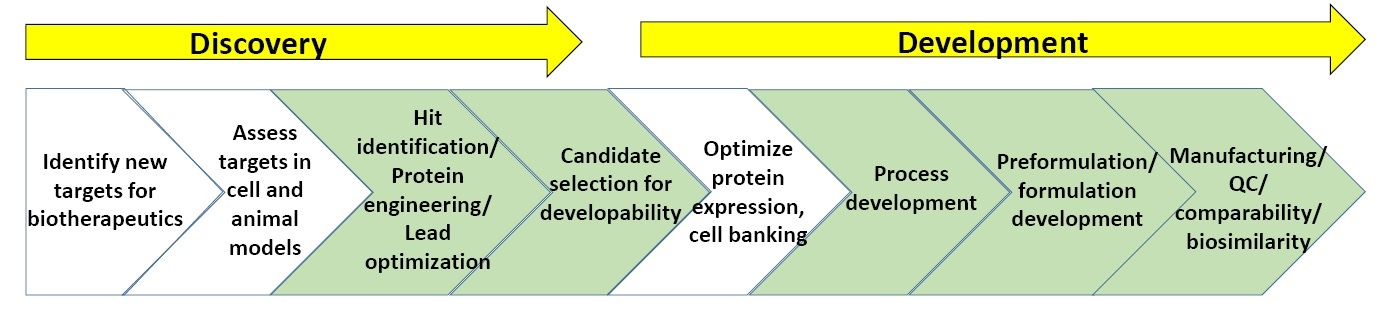
Figure 3 : Système général de procédés dans la recherche et le développement de composés biopharmaceutiques.
La figure 3 révèle un système généralisé dans la recherche et le développement de composés biopharmaceutiques. Les parties vertes mettent en évidence les points pour lesquels la caractérisation biophysique, notamment les tests de stabilité, sont les plus couramment utilisés. À la fin de cette article, une liste de lecture sur la recherche et le développement de procédés biopharmaceutiques vous est proposée.
La séquence d’acides aminés, ou structure primaire, est un élément de base de la chaîne polypeptidique et de la structure de la protéine. Il est important de comprendre et de caractériser la structure tridimensionnelle de la protéine, également appelée structure d’ordre supérieur. Il existe 3 niveaux de structure d’ordre supérieur de la protéine : la structure secondaire de la protéine qui décrit le repliement local de la structure primaire de la protéine, notamment l'hélice α et le feuillet β,la structure tertiaire qui correspond à la structure tridimensionnelle d’une protéine, provenant d’un ensemble d'éléments de la structure secondaire, et la structure quaternaire qui regroupe l’association d’au moins deux chaînes polypeptidiques identiques ou différentes.
En tenant compte de cela et pour créer des produits biopharmaceutisues bénéfiques, les scientifiques cherchent d’abord des molécules présentant une stabilité élevée pendant la sélection du candidat et pensent qu’une stabilité accrue peut être apportée par ingénierie génétique. La purification des protéines est un processus destiné à isoler la protéine d’un milieu dans lequel elle est stable, correctement pliée et active. Il est donc primordial d'utiliser à bon escient les solutions tampon, les additifs, les techniques de purification et les conditions de conservation afin de maintenir une protéine aussi stable que possible à ce stade.
Lorsque les molécules de protéines sont exposées à des facteurs de contrainte fréquemment présents lors de la formulation et de la production de procédés biopharmaceutiques comme la chaleur, les produits chimiques, les variations de pH, la pression, cela peut conduire à la dénaturation de la protéine. Les protéines-médicaments administrées par voie sous-cutanée doivent être stables et résistantes à des concentrations très élevées en protéine (plus de 100 mg/ml), que ce soit dans un flacon ou une seringue pré-remplie, et cela pendant plusieurs années. Les protéines en solution risquent également de subir des modifications telles que la déamidation et l’oxidation pouvant donner lieu à des protéines inactives, dénaturées.
Dans le cas de procédés biopharmaceutiques protéiniques, la dénaturation ou d’autres modifications pourraient entraîner la formation d'agrégats susceptibles de réduire son efficacité ou de lui conférer des propriétés non-fonctionnelles. Il est peut-être encore plus important de souligner que l'agrégation de protéine peut provoquer des réponses indésirables et immunogènes potentiellement mortelles chez certains patients. L’utilisation de protéines intrinsèquement stables permet de fabriquer de manière plus avantageuse des produits médicamenteux efficaces, bénéfiques et sans danger.
La DSC permet d’analyser la stabilité conformationnelle d’une molécule et les modifications de la structure tertiaire et quaternaire qui surviennent lorsqu’une protéine est dénaturée sous l’effet de la chaleur mais elle permet également de mesurer l’effet des facteurs intrinsèques et extrinsèques sur la stabilité protéique. La DSC est considérée comme la meilleure et la plus performante méthode d’analyse quantitative capable de déterminer la stabilité thermique de protéines biopharmaceutiques. De plus, elle est utilisée comme un bon indicateur de stabilité à long terme[1,10-14]. La DSC permet de générer des valeurs T M qui est un paramètre fréquemment utilisé pour évaluer le niveau de stabilité lors du processus de sélection du produit candidat (potentiel de développement), lors de l’analyse de la formulation et lors du développement de procédés. La plupart des protéines stables ont une TMélevée. L’enthalpie (∆H), les valeurs de Tonset, de T1/2 et de ∆Cp mesurées par DSC sont aussi utilisées pour évaluer le niveau de stabilité et valider les données de DSC, l’analyse quantitative de la protéine dénaturée et la structure d’ordre supérieur « cartographie peptidique »[10-14].
L’analyse réalisée par DSC de protéines présentes dans des conditions définies est une analyse quantitative qui peut être reproduite si les protéines analysées sont identiques ou hautement similaires.(Figure 4). En d’autres termes, les thermogrammes DSC présenteront un profil reproductible et les paramètres (notamment les valeurs TM, ΔH et Tonset) seront jugés acceptables[12-14]. Si l’on observe des thermogrammes et des paramètres de DSC différents, cela semblera indiquer qu’il y a eu par exemple soit un repliement anormal de protéines, un phénomène de dégradation, d’agrégation, des écarts observés dans la solution tampon, des changements dans la modification post-translationnelle ou d’autres modifications de la structure d'ordre supérieur, ayant tous une incidence négative sur la stabilité conformationnelle de la protéine.
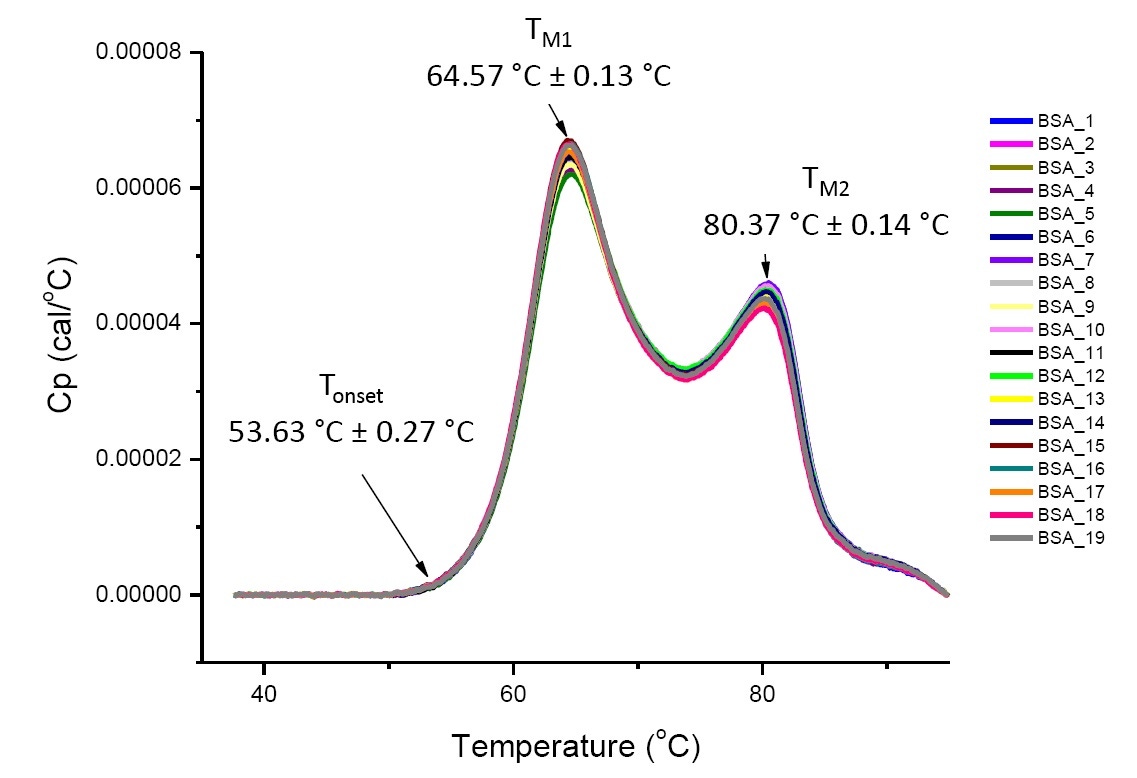
Figure 4. Représentation de 19 thermogrammes DSC d’albumine de sérum bovin (Sigma A1933, purifié par chromatographie) dans une solution tampon PBS. Les données de DSC qui apparaissent ont été déterminées suite à la normalisation de la fréquence de balayage, au passage d’une solution tampon à une autre et à la suppression d’une solution tampon. La moyenne et l’écart-type des valeurs Tonset, TM1, et TM2 sont indiqués.
Les résultats reproductibles et quantitatifs font de la DSC un outil fiable capable de déterminer la structure d’ordre supérieur lorsqu’il s’agit d’évaluer un produit pendant sa fabrication (comparabilité par lot et par site), de comparer les variantes protéiques et les produits modifiés (modifications de structure suite à une glycosilation, déamidation ou oxydation) et également lorsqu’il s’agit de biosimilarité. Les données de DSC figurent également dans les documents relatifs au normes en tant qu’éléments importants pour déterminer la structure d’ordre supérieur indispensable à la fabrication de nouveaux procédés et de biosimilaires. Lors d’une enquête réalisée par des chercheurs en technologie biopharmaceutique, la DSC a été définie comme étant une technique d’analyse très utile voire même extrêmement utile pendant la phase de sélection du produit candidat, de développement de la formulation, de caractérisation du produit et lorsque l’on parle de comparabilité et de biosimilarité[15].
Les thermogrammes DSC révèlent l’existence de plusieurs transitions lorsqu’il s’agit de protéines mutidomaines (anticorps) dites dénaturées (Figure 2). La DSC est capable d’identifier et de quantifier les différents domaines, elle peut déterminer les valeurs TM pour plusieurs transitions (deux, trois ou plus). Les valeurs TM peuvent être déterminées de manière très simple à partir des pics retrouvés sur les thermogrammes DSC et sans avoir recours à une analyse complexe des données. Il existe d’autres tests biophysiques capables de déterminer la TM tels que le dichroïsme circulaire (DC), la fluorimétrie différentielle à balayage (DSF), et la fluorescence intrinsèque (IF) mais ils peuvent uniquement mesurer la première TM (rencontrée pour les températures les plus basses) ou la TM la plus prépondérante pour des protéines multidomaines. L’extraction de plusieurs données spectroscopiques ou de fluorescence nécessite une adéquation complexe des données et peut ne pas être reproductible.
Par rapport à d’autres techniques d’analyse de la TM, la DSC nécessite souvent plus d’échantillons de protéines par test et peut avoir une capacité d’analyse inférieure. Si l’échantillon est limité, il existe une solution qui consisterait tout d’abord à réaliser un classement par ordre de grandeur des TM soit par DSF ou IF puis de sélectionner plusieurs échantillons afin de valider la TM par DSC. Il est important que cette étape de validation par DSC soit réalisée et que les techniques de fluorescence ou de spectroscopie ne soient pas considérées comme les seules à pouvoir mesurer la TM lors des tests de stabilité. Les tests qui utilisent souvent la fluorescence connaissent des artéfacts qui brouillent les mesures et modifient les TM pouvant passer d’une valeur très basse à très haute et inversement. Certaines protéines et solutions tampon ne sont pas compatibles avec la fluorescence, c’est pour cela que les différences de TM ne sont parfois pas détectées. En définitive, la fluorescence et la spectroscopie sont incapables de mesurer l’enthalpie calorimétrique et de déterminer d’autres paramètres thermodynamiques, contrairement à la DSC.
La DSC qui est capable de déterminer la stabilité thermique est réputée être un test d’excellence par les chercheurs du secteur biopharmaceutique car cette technique :
Dans cet article mais également dans d’autres publications, demandes de notification du laboratoire Malvern, vous trouverez des exemples expliquant la façon dont les données DSC sont utilisées dans le développement et la recherche biopharmaceutique.
Lors de la phase de recherche permettant d’identifier une protéine biopharmaceutique, le but est de détecter l’interaction du médicament avec sa cible. Une fois la cible identifiée, il faut alors trouver un médicament capable d'interagir avec cette cible, ce qui permettra d’obtenir l’effet biologique escompté entraînant un résultatclinique favorable, à savoir la réduction ou l’élimination d’un état pathologique. Après avoir identifié les candidats-médicaments, il est important d’évaluer le « potentiel de développement » de ces candidats biopharmaceutiques.
Le potentiel de développement d’une protéine-médicament est une évaluation des propriétés biophysiques de la protéine visant à déterminer la capacité d’une protéine à résister aux facteurs de contrainte rencontrés lors de la production, de la purification, de la formulation, du conditionnement, du transport et de la conservation à long terme (stabilité) de cellules en culture. Il est important de connaître :
Il est important d’analyser le potentiel de développement d’une protéine médicament candidate à la fin de la « recherche » avant que le développement pré-clinique ne débute, car une fois que la protéine candidate est sélectionnée et que l’étude portant sur la capacité de production est amorcée, toute tentative visant à modifier le médicament ou le processus entraînera une hausse des coûts et vraisemblablement l'abandon du projet. Le fait de prendre de bonnes décisions à ce stade se traduit par moins d’échecs et par une réduction significative des coûts pour la société pharmaceutique et pour la production de médicaments performants et peu onéreux.
À ce stade avancé de la découverte ou précoce du développement, les tests biophysiques disposent souvent d’une quantité limitée de protéines purifiées et les tests de potentiel de développement d’un temps limité. Les méthodes biophysiques telles que la fluorescence, la diffusion de la lumière, la chromatographie d'exclusion stérique et la DSC sont toutes utilisées fréquemment à ce stade. D’autres tests comme l’ultracentrifugation analytique et l’échange hydrogène/deutérium associés à la spectrométrie de masse peuvent fournir d’importantes informations.
Le profilage simple et précoce de protéines-médicament utilise la DSC pour classer dans l’ordre les valeurs de TM et peut être très utile pour comparer les candidats médicaments afin de trouver les protéines possédant une stabilité optimale, pour estimer la stabilité à long terme et pour éviter que ces molécules aient moins de structures thermiquement stables. Les protéines ayant des valeurs de TM les plus basses sont les moins intéressantes pour devenir des candidats médicaments, en raison de leur plus grande instabilité face à la température.
Doyle et al.[16] ont publié une étude de cas faisant appel à des méthodes biophysiques pour caractériser les candidats bispécifiques à l’adnectine. Les test biophysiques comprenaient la chromatographie d'exclusion stérique (SEC), l'ultrafiltration à flux tangentiel, la chromatographie d’exclusion stérique (filtration sur gel) couplée avec la mesure de la diffusion de la lumière multi-angle (SEC-MALS), la cristallographie aux rayons X, la résonance plasmonique de surface (SPR), la fluorimétrie différentielle à balayage (DSF) et la calorimétrie différentielle à balayage (DSC). La comparaison de deux candidats d’adnectine par DSC a montré que ce candidat B affichait une TM et une Tonset plus élevée (les deux indiquant une plus grande stabilité thermique) par rapport au candidat A. A l’aide de la DSC, on s’est aperçu que le candidat B montrait également des signes de dénaturation réversible, il faut savoir que l’échantillon de protéine a été refroidi puis réchauffé lors de la DSC et que le 2émecycle de chauffe présentait le même profil DSC. Ce caractère réversible indique également des niveaux d’expression accrus et une tendance à la formation d’agrégats moins marquée.
Les anticorps monoclonaux font partie de la plus grande catégorie de protéines contenues dans les produits biopharmaceutiques et présente sur le marché ou lors des essais cliniques. Les anticorps monoclonaux présentent en général un thermogramme complexe de protéines multidomaines lors de la DSC. Le pic le plus élevé, le plus proéminent correspond au fragment Fab de l’anticorps. Les domaines CH2 et/ou CH3 sont aussi fréquemment représentés. Les valeurs respectives et les TM des différents fragments dépendent d’anticorps monoclonaux spécifiques et peuvent varier selon les sous-catégories et les techniques, comme on peut le constater dans les exemples ci-dessous.
En 2017, Garber et Demarest ont publié un article expliquant l’utilisation de la DSC pour caractériser 17 anticorps thérapeutiques présentant des séquences variables de pleine longueur[17,18]. Grâce à l’utilisation du DSC MicroCal VP-Capillary, on a observé que les fragments Fab de ces anticorps présentaient des zones de transitions relatives à la dénaturation avec des points médians de TM variant de 57°C à 82°C. Les auteurs ont démontré que l’IgG1 faisait partie de la sous-classe la plus stable d'immunoglobuline humaine, suivie par l’IgG2 et l’IgG4, et ont constaté que les variations de stabilité entre anticorps de même sous-classe provenaient des domaines variables. Les caractéristiques propres à chaque gène du facteur V sont liées à la stabilité du fragment Fab.
Certains fragments Fab de l’anticorps peuvent être extrêmement stables, tandis que d’autres sont relativement instables. Comme on a pu le constater pour plusieurs hybrides, la très faible stabilité du fragment Fab observée à partir de la TM était liée à une faible expression de l'anticorps et à des problèmes accrus d’agrégation par rapport aux anticorps présentant une plus grande stabilité. Par conséquent, il est plus difficile de purifier une protéine avec une faible TM, ce qui se traduit par une baisse de rendement pendant le développement de procédés et par une supposée augmentation de la formation d’agrégats pendant la conservation. Les données recueillies lors de cette étude[17] ont prouvé que les données relatives à la stabilité et obtenues par DSC sont des éléments utiles et nécessaires à la sélection du candidat pour les médicaments biopharmaceutiques.
Ionescu et al.[19] ont crée des profils DSC (utilisant le DSC MicroCal VP-Capillary D) de trois anticorps monoclonaux humanisés de type IgG1 et de leurs fragments Fab et Fc à pH neutre. Hormis quelques exceptions, les thermogrammes ont montré que la transition présentant la plus grande valeur expérimentale d’enthalpie contenait les fragments Fab (similaire aux résultats indiqués dans la référence 17). Les TM mises en évidence présentaient également d’importantes variations, même pour les fragments Fab provenant de la même lignée germinale. Ionescu at al. ont proposé d'utiliser l’enthalpie de dénaturation comme paramètre clé pour reconnaître les éventuelles dénaturations pouvant apparaître dans le profil de fusion d’un anticorps IgG1 intact. Certains thermogrammes DSC ont révélé l’existence de deux transitions : la première représentant la dénaturation du fragment Fab et du domaine CH2, et la seconde correspondant à la la dénaturation du domaine CH3. Pour d’autres anticorps, la première transition représente la dénaturation du domaine CH2 et la seconde la dénaturation du fragment Fab et du domaine CH3. Dans d’autres cas, le profil DSC présentait 3 transitions pendant lesquelles la dénaturation du fragment Fab avait lieu à différentes températures par rapport à la fusion des domaines CH2 et CH3 . La faible stabilité ou hétérogénéité du fragment Fab peut s’avérer problématique pour la conservation à long terme ou la constance de la production. Les auteurs ont estimé qu'il était important de bien comprendre les caractéristiques du profil DSC afin de pouvoir sélectionner le candidat dès le début du développement des anticorps monoclonaux thérapeutiques.
Le travail décrit dans les deux articles précédents mais également dans d’autres publications au cours de la même période a servi de base aux études de cas qui ont suivi, aux études permettant d’évaluer la stabilité et a permis de comprendre l'utilisation de la DSC faisant partie intégrante de la sélection du candidat biopharmaceutique.
Pour définir plus précisément la stabilité thermique de candidats médicaments, les protéines sont fréquemment analysées à des valeurs de pH différentes pour voir s’il existe des différencses. Ces différents pH peuvent correspondre à des valeurs retrouvées dans la formulation et/ou dans les solutions tampons utilisées lors de la fabrication du médicament.
Jiang et al.[20] ont publié une étude de cas reposant sur la caractérisation de la structure d'ordre supérieur pour évaluer le potentiel de développement (capacité de fabriquer et de préserver la qualité finale du produit) de deux anticorps-médicaments. Les anticorps X et Y étaient des anticorps monoclonaux IgG2 dirigés contre la même cible et qui affichaient des activités biologiques similaires. Grâce à l’utilisation de plusieurs techniques dont la DSC, la mesure du dichroïsme circulaire dans l’UV proche, la spectroscopie infrarouge à transformée de Fourier, la diffusion dynamique de la lumière, la spectroscopie de fluorescence et la chromatographie d’exclusion stérique, ils ont pu analyser :
Des milieux acides ont été sélectionnés pour reproduire les conditions tampons utilisées pendant la purification de l’anticorps et l’inactivation virale due à cet anticorps. Les tampons de pH 5 simulaient des conditions de conservation (formulation) et ceux de pH 7 étaient utilisés tout au long de la purification.
Les images obtenues de deux anticorps candidats présents dans une solution tampon PBS de pH 7 grâce à l’utilisation du système DSC MicroCal VP-Capillary ont révélé l’existence de deux transitions : le domaine CH2 et le fragment Fab correspondaient à la première transition tandis que la dénaturation du domaine CH était représentée par la seconde transition[20]. À partir des valeurs plus élevées de TM et de Tonset obtenues par DSC, on s’est rendu compte que l’anticorps monoclonal Y était légèrement plus stable d’un point de vue thermique à un pH 7. Pour le tampon de pH 3 (C3N), on a relevé qu’une seule transition thermique, ce qui indique que les échantillons étaient déjà partiellement dénaturés à la température initiale, en raison du faible pH. Comme on pouvait s’y attendre, les températures de transitions thermiques et d’enthalpie de dénaturation des deux candidats présents dans un tampon d’acide cyanurique de pH 3 étaient nettement inférieures à celles retrouvées dans la solution tampon PBS. Toutefois, l’anticorps monoclonal X (TM de 63,2°C) présentait une meilleure stabilité thermique que l'anticorps monoclonal Y (TM de 48,2°C) présent dans le tampon d’acide cyanurique, même si ce denier était plus stable d’un point de vue thermique à un pH neutre. Bien que ce résultat soit atypique, il est cohérent avec les données recueillies par la mesure du dichroïsme circulaire dans l’UV proche qui ont montré que l’anticorps monoclonal X conservait davantage sa structure tertiaire à un pH 3. Les variations de stabilité thermique des deux candidats induites par le traitement à pH 3 semblaient être complètement réversibles après avoir réalisé la dialyse dans la solution tampon PBS.
Par ailleurs, les résultats obtenus par DSC ont indiqué que les deux protéines étaient très stables à pH neutre et que l’anticorps monoclonal Y présentait une meilleure stabilité que l’anticorps monoclonal X dans la solution tampon PBS mais avec une plus faible stabilité que celle de l’anticorps monoclonal X dans la solution d’acide cyanurique[20]. Les données obtenues par dichroïsme circulaire et DSC ont indiqué que l’anticorps monoclonal Y connaissait davantage de modifications de sa structure à pH 3 que l’anticorps monoclonal X. Les variations de stabilité thermique induites à pH 3 étaient complètement réversibles pour les deux anticorps monoclonaux, même si les modifications de la structure tertiaire semblaient être partiellement irréversibles pour l’anticorps monoclonal Y. Par rapport à l’anticorps monoclonal X, l’anticorps monoclonal Y présentait une meilleure stabilité thermique et une meilleure stabilité à la conservation à une température de 37°C lors de sa formulation tampon à pH 5. Globalement, les résultats indiquent qu’il existe différentes forces conductrices nécessaires à la stabilité du produit (résistance aux variations de pH) et à la stabilité lors de la conservation (incidence d’une température élevée).
L’anticorps monoclonal Y a pu poursuivre sa phase de développement plus approfondie puisqu’il présentait une meilleure stabilité thermique dans la solution tampon PBS à pH 7 et une meilleure stabilité à pH 5 lors de la phase de conservation à 37°C[20]. De plus, en raison de l’irréversibilité de dégradation de la structure tertiaire et de l’auto-association observée après incubation à pH faible dans la solution tampon PBS, la recommandation visant à faire progresser l’anticorps monoclonal Y s’est vue être accompagnée d’une recommandation visant à développer le processus de purification afin de minimiser l’exposition de l’anticorps monoclonal Y à un pH 3. Cette étude de cas a démontré à quel point les données de stabilité étaient utiles pour pouvoir réduire au maximum la quantité d’agrégats irréversibles et de variants conformationnels produits, et pour accroître le rendement du processus[20].
Plusieurs autres études de cas utilisant la stabilité thermique pour évaluer et prédire la développabilté sont résumées dans l’article publié par Satish et al.[21]
Tavakoli-Kenshe et al.[22] ont voulu voir quels autres facteurs outre les études de stabilité pourraient être utilisés pour prédire le potentiel de développement. Ils ont étudié 5 variants d’anticorps IgG1 et IgG4 et utilisé un rhéomètre rotatif capable de cisailler dans des conditions définies l’interface air-liquide pour mesurer la stabilité présente dans ce milieu. Les anticorps ont été analysés pour évaluer la stabilité selon le rendement du rhéomètre puis comparés aux données de stabilité thermique accélérée et à la température de fusion TM1 du domaine CH2 obtenues grâce au système DSC MicroCal VP-Capillary pour étudier la complémentarité des techniques. Les résultats semblent indiquer que les techniques sont orthogonales, appliquées avec des méthodes thermiques basées sur l'interaction intramoléculaire et que la stabilité mesurée par le rhéomètre reposait sur la localisation de la dénaturation. Cela nous permet d’affirmer que des régions moins stables favorisent le phénomène d'agrégation. La modélisation moléculaire permet d’observer l’incidence des modifications sur la structure de l’anticorps et de reconnaître l'implication de la structure fonctionnelle du fragment Fab et Fc dans la détermination de la stabilité de la protéine. Les données présentent l’essai de cisaillement comme étant un potentiel indicateur de stabilité orthogonale et comme complémentaire aux méthodes thermiques traditionnelles telles que la DSC permettant de sélectionner la protéine candidate la plus stable pour l’étape de purification.
En général, dans la recherche biopharmaceutique, les molécules « hit » ou les molécules thérapeutiques « parentales » sont modifiées ou conçues pour améliorer leurs caractéristiques biophysiques et permettre à la protéine de mieux se développer. Après la conception mais avant la sélection du candidat, il est important d’évaluer les différentes protéines conçues en termes de stabilité pour voir si cela n'entraîne pas d’effets nuisibles. Un exemple a été présenté dans une demande de notification du laboratoire Malvern[23], dans lequel le système DSC MicroCal VP-Capillary était utilisé comme instrument d’analyse de l’anticorps parent et de deux anticorps produits par génie génétique pour pouvoir prédire la stabilité thermique et le potentiel de développement d’hybrides issus du génie génétique. La formation d’agrégats (observée par chromatographie d’exclusion stérique) apparue après conservation accélérée a également été étudiée. L’ anticorps issu du génie génétique présentant la plus importante baisse de TM corrélée à l’anticorps hybride comporte la plus grande quantité d’agrégats après avoir analysé la stabilité accélérée, permettant ainsi de contribuer à la sélection du candidat.
Demarest et al. ont produit un fragment Fab instable et utilisé le DSC MicroCal VP-Capillary pour démontrer que la stabilité de la protéine était améliorée[18, 24]. Pour essayer de développer des stratégies en vue de stabiliser l’anticorps et le fragment de l’anticorps, il ont sélectionné un fragment Fab imprévisible capable de reconnaître la toxine tétanique isolée à l’origine d’une source humaine et ont réalisé une mutagénèse pour recréer de la stabilité dans le fragment Fab. Les auteurs ont sélectionné 45 positions de résidus pour la randomisation et près de 4 500 colonies différentes ont été choisies et mises en culture. Les surnageants contenant le fragment Fab ont été chauffés à trois températures différentes (70°C, 72°C et 74°C). Les variants indiquant une meilleure thermostabilité ont été soumis au test de thermotolérance pour confirmer leurs propriétés (voir la référence 24 pour obtenir la liste complète des protéines mutées neutralisantes). La thermostabilité a été améliorée pour environ 1 % des variants de la bibliothèque. Quatorze des « touches » concernaient le domaine VH, les quatre autres concernaient le domaine VL. Ce résultat suggère que la stabilité du fragment Fab natif était limitée par la stabilité marginale de la région variable VH. De façon surprenante, aucune protéine mutée de la bibliothèque de domaines constants d'environ 2 000 membres ne s'est avérée stabiliser le fragment Fab dans son ensemble. Les auteurs supposent que des mutations ont vraiment lieu au sein des régions constantes mais que la stabilité limitée de la région variable VH contrôle la température à laquelle le fragment Fab se dénature et réduit considérablement les possibilités d’observer de tels événements.
Quatre protéines mutées neutralisantes ont été découvertes au sein de la région variable VL de la toxine tétanique[24]. La mutation du résidu consensus VK4 W50 en Ala (le résidu consensus pour VK1) ou His était fortement stabilisatrice. L'histidine se trouve rarement en position 50 dans les domaines variables kappa humains, mais se trouve souvent dans les domaines variables lambda humains. Ce résidu est proche de l'interface entre les domaines VH/VL. Les auteurs ont émis l’hypothèse selon laquelle sa contribution à la stabilité du fragment Fab pourrait être liée au renfort potentiel du domaine VH puisque ce domaine VH en particulier semble limiter la stabilité du fragment Fab de la toxine tétanique. Néanmoins, des études s'intéressant à la région variable VL isolée ont suggéré le contraire.
Douze hybrides ont été générés, contenant entre trois et onze protéines mutées neutralisantes ayant été identifiées lors du criblage initial[24]. Différentes combinaisons ont été dérivées de façon rationnelle pour déterminer la part de responsabilité de chaque protéine mutée dans la stabilisation du fragment Fab. Les multiples mutations créées au niveau du fragment Fab avec la toxine tétanique ont permis d’améliorer l’expression du fragment Fab lors de transformation parallèle ou d’expérience d’expression, ce qui laisse à penser que les hybrides les plus stables sont capables d'accroître l’expression dans les cellules. Les meilleurs hybrides présentaient systématiquement une activité au moins trois fois supérieure à l'expression obtenue avec le type sauvage. La stabilité de chaque fragment Fab était évaluée par DSC et dichroïsme circulaire (DC). Les valeurs TM mesurées étaient utilisées pour évaluer le niveau de stabilité du fragment Fab.
Les potentielles conséquences que ces mutations pourraient avoir sur la fixation du fragment Fab sur l’antigène constituent un des facteurs importants à prendre en considération. Les mutations thermostabilisatrices ont été déterminées à partir du criblage initial de la méthode immuno-enzymatique ELISA à visée quantitative, capable de détecter à la fois le domaine CL et le marqueur histidine au niveau de l'extrémité C du domaine CH1. Les auteurs se sont aperçus, grâce à l’utilisation de la méthode immuno-enzymatique ELISA, que les mutations thermostabilisatrices permettaient d’améliorer la constante d’affinité du fragment Fab avec la toxine tétanique par rapport au type sauvage[24]. La capacité fonctionnelle de chaque variant du fragment Fab semble être directement corrélée à sa stabilité, comme le décrit la TM obtenue par DSC. Le fait de pouvoir corréler l’activité fonctionnelle et l’expression de la protéine à la TM permettra de sélectionner l’hybride le plus stable et par conséquent d’aboutir à un meilleur potentiel de développement.
Seeliger et al.[25] ont prévu des stratégies computationnelles de conception pour modifier systématiquement un anticorps ayant tendance à s’agréger in vitro. L'étroite relation qui existe entre un anticorps issu de l'ingénierie génétique et un anticorps monoclonal de type sauvage (mAb1) a permis d’observer des niveaux de stabilité supérieurs, définis par des méthodes biophysiques (notamment grâce au DSC MicroCal VP-Capillary) et des tests de stabilité à long terme. Comme l’ont expliqué Demarest et al dans l’exemple cité ci-dessus concernant le fragment Fab et la toxine tétanique[24], les niveaux d’expression des protéines mutées ont également progressé par rapport au candidat de type sauvage. Les données expérimentales et informatiques conjointement utilisées lors de cette étude témoignent de l’importance de l’utilité de ces méthodes computationnelles visant à optimiser les performances de l’anticorps pour améliorer la stabilité.
L’analyse de la protéine d’origine de type sauvage a enregistré une TM de 68°C pour la première transition du thermogramme (identifiée comme étant le fragment Fab). Lorsque l’on examine les thermogrammes DSC correspondants aux mutations de la chaîne lourde ou légère, on observe que la TM de la première transition thermique (Fab) est passée de 68,9°C à 72,8°C. Au niveau de l’anticorps constitué de chaines lourdes et légères issues du génie génétique, on observe un premier endotherme à 70,5°C avec un pic très marqué (Fab) à 83,5°C. La TM relative à la dénaturation du fragment Fab de l’anticorps augmente d’environ 16 K après avoir supprimé les éventuels « risques » pouvant apparaître au niveau de la chaine lourde et légère. Lorsque l’on enregistre une dénaturation sous l’effet de variations au niveau de fluorophores tels que le tryptophane, on observe le même phénomène[25].
La simulation par ordinateur avait anticipé le fait que la plupart des mutations permettrait d’augmenter la stabilité thermodynamique. Ces résultats concordaient avec ceux obtenus à partir des mesures de DSC et de diffusion de la lumière à angle droit (RALS). Les deux essais ont indiqué que le niveau de stabilité ne cessait d’augmenter avec un nombre croissant de mutations. À partir de ces deux essais, les auteurs ont aussi constaté que la chaine légère issue du génie génétique avait un effet notable sur la stabilité, ce qui coïncide avec les variations de thermostabilité calculées pour chaque mutation[25].
Tous les variants de l’anticorps monoclonal mAb1 issus du génie génétique étaient exprimés avec des titres de plus de 50 mg/L, tandis qu’avec le type sauvage, les titres de l’anticorps monoclonal mAb1 étaient inférieurs à 1 mg/L. L’augmentation spectaculaire des niveaux d’expression des variants comportant uniquement une chaine issue du génie génétique n’était pas prévue d’après les résultats de DSC, on a donc observé une légère augmentation du niveau de stabilité pour ces variants. Cependant, les calculs semblent indiquer que les différents domaines de l’immunoglobuline sont plus stables lorsque des mutations sont provoquées. Lors des essais faisant intervenir un traitement thermique, il est possible que le niveau de stabilité de chaque chaine soit le facteur limitant, ce qui permet de préciser que les effets observés étaient plutôt des effets cinétiques que des effets liés à l’équilibre thermodynamique. Dès qu’un domaine se dénature, l’effet stabilisateur de la formation du complexe disparaît et, par conséquent, la seconde chaine commence également à se dénaturer. En ce qui concerne la production et le repliement in vivo de protéines, la présence d’un domaine stable à l’état natif servant de point de nucléation pour une chaine moins stable pourrait suffire à augmenter considérablement la quantité d’anticorps se trouvant dans leur état natif[25].
Du point de vue d’une société capable de développer un médicament biopharmaceutique, est-ce qu’une protéine qui devient stable progressivement peut induire une meilleure stabilité à long terme et une meilleure durée de conservation ? Les auteurs ont mené une étude accélérée de stabilité à 40°C avec des variants issus du génie génétique et ont constaté une amélioration considérable de la stabilité à long terme comme en témoigne la teneur en monomère déterminée par SEC. Les variants hybrides issus du génie génétique étaient également les plus stables pendant les 40 jours de test[23]. La stabilité thermodynamique des domaines variables est sans aucun doute un facteur important pour la stabilité à long terme. Les données énoncées par l’auteur ainsi que les autres données publiées révèlent clairement que la stabilité thermodynamique et la durée de conservation ne sont pas obligatoirement corrélées et qu’il pourrait s’avérer nécessaire de procéder à des modifications en vue d’améliorer la stabilité lors de la phase de formulation [25,26,27,28].
De récentes publications s'intéressent à l’ingénierie rationnelle, à l'utilisation de la DSC et à d’autres instruments biophysiques capables de caractériser la stabilité d’une protéine, dont notamment :
Un anticorps monoclonal a été analysé au National Institute of Standards and Technology (NIST) [35] pour connaître son potentiel de développement avec des instruments biophysiques permettant de déterminer la structure d’ordre supérieur et généralement utilisés pour valider un anticorps monoclonal comme candidat-médicament. L’anticorps monoclonal présent dans cet institut de recherche ne prétend pas être une molécule thérapeutique et l’évaluation du potentiel de développement de cet anticorps monoclonal a été réalisée comme si on s'intéressait à un anticorps IgG1 spécifique à une certaine classe[36]. Cet anticorps a été analysé dans différentes solutions tampon par électrophorèse, par chromatographie d’exclusion stérique (filtration sur gel) couplée avec la mesure de la diffusion de la lumière multi-angle (SEC-MALS), par diffusion dynamique de la lumière (DLS) et par DSC. La stabilité thermique de l’échantillon de cet anticorps monoclonal présent dans une solution tampon PBS a été déterminée par le DSC MicroCal VP-Capillary. L’échantillon du NIST comporte trois principales transitions thermiques qui correspondent au domaine CH2 à 71,2°C, au domaine CH3 à 84,1°C et au fragment Fab à 88,9°C.
Par rapport aux profils DSC d’immunoglobulines IgG1 figurant dans la référence 17, le fragment Fab analysé par le NIST est plus stable à 88,9°C, on en a donc conclut que la molécule présentait un profil de structure d’ordre supérieur favorable[36]. D’après l'ensemble des données exhaustives d’analyse biophysique que nous avons en notre possession, l’anticorps monoclonal du NIST présente un profil de potentiel de développement favorable et les modifications observées dans la molécule dues aux conditions de contrainte ne sont pas considérées comme un facteur de risque pouvant être atténué grâce à la bonne formulation et à la production plus tard au cours du processus de développement.
Les résultats présentés dans cet article illustrent clairement l’importance et l’efficacité d’intégrer la DSC en tant que test biophysique de stabilité pendant la phase de sélection du candidat biothérapeutique. L’interprétation des résultats issus de la DSC et d’autres tests de stabilité a permis aux sociétés biopharmaceutiques de prendre des décisions éclairées sur les candidats médicaments les plus stables, ce qui signifie que certains pourront atteindre la phase de production et de purification et que d’autres moins susceptibles de présenter une stabilité à long terme et des problèmes d'agrégation pourront passer à la phase de formulation finale et devenir par la suite un médicament. Cela se traduit par une production plus efficace et peu onéreuse de médicaments, et par une probabilité accrue de voir la formulation médicamenteuse finale rester active, stable et sous sa forme native correctement repliée.
Analytical Techniques for Biopharmaceutical Development, R. Rodriguez-Diaz, T. Wehr, S. Tuck (eds.), Taylor & Francis, New York USA (2005).
Biophysical Characterization of Proteins in Developing Biopharmaceuticals, D.J. Houde, S.A. Berkowitz (eds.), Elsevier, Amsterdam, Netherlands (2015).
Biophysical Methods for Biotherapeutics: Discovery and Development Applications, T.K. Das (ed.) John Wiley & Sons, Hoboken NJ USA (2014).
Biophysics for Therapeutic Protein Development, L.O. Nahri (ed.), Springer New York, USA (2013).
State-of-the-Art and Emerging Technologies for Therapeutic Monoclonal Antibody Characterization Volume 1. Monoclonal Antibody Therapeutics: Structure, Function, and Regulatory Space, J.E. Schiel, D.D. Davis, O.V. Borisov (eds.), ACS Symposium Series Vol 1176 (2014). doi: 10.1021/bk-2014-1176.
State-of-the-Art and Emerging Technologies for Therapeutic Monoclonal Antibody Characterization Volume 2. Biopharmaceutical Characterization: The NISTmAb Case Study, J.E. Schiel, D.D. Davis, O.V. Borisov (eds.), ACS Symposium Series Vol 1201 (2015). doi: 10.1021/bk-2015-1201.
State-of-the-Art and Emerging Technologies for Therapeutic Monoclonal Antibody Characterization Volume 3. Defining the Next Generation of Analytical and Biophysical Techniques, J.E. Schiel, D.D. Davis, O.V. Borisov (eds.), ACS Symposium Series Vol 1202 (2015) DOI: 10.1021/bk-2015-1202.